近年来,以深度卷积神经网络为代表的深度学习方法通过对大量的标注样本进行学习,能够自动提取出大量有效的高层次特征,从而提高肝脏分割精度. 全卷积神经网络[12-13]可以直接处理整个图像,实现端到端的图像分割. 2015年,Ronneberger等[14]提出UNet并将其应用于生物医学图像分割领域中,这是其在生物医学图像分割领域的第1次应用. 由于UNet可以结合高级语义信息和低级信息,近几年来国内外很多研究者将UNet作为主干网络应用到很多自动肝脏分割任务中. Cicek等[15]将UNet拓展到三维图像领域. Christ等[16]采用2个级联的UNet模型实现肝脏及肿瘤的分割. 刘哲等[17]将改进的UNet与主动轮廓边界演化方法相结合实现肝脏CT图像的精确分割.
随着研究的不断深入,有学者发现在使用3D UNet进行医学图像分割时会出现特征冗余现象. 注意力机制可以增加有效特征的权重,使得网络忽略无关信息并关注有效信息[18],提高深度网络学习图像特征的能力. Oktay等[19]在UNet的每个跳跃连接层的末端使用一个注意力门控信号,控制不同空间位置处特征的重要性. Hu等[20]提出另一种注意力机制,即挤压和激发网络(squeeze-and-excitation networks,SENet),通过显式建模特征通道之间的相互依赖关系,自适应地重新校准通道方向的特征响应,提升有效特征并抑制无关特征. Roy等[21]受SENet的启发提出并行的空间/信道挤压和激励模块(concurrent spatial and channel squeeze and excitation block,scSE-block),在CT器官分割上取得了可观的效果. 本研究在scSE-block的基础上进行改进,通过并行地加入全局最大池化层得到scSE-block+,并将其嵌入3D UNet构成3D scSE-UNet分割网络. 该网络通过同时关注特征图的通道域和空间域,强化有效特征信息并抑制冗余信息,得到更为有效的特征,有助于进一步提升肝脏分割精度.
目前使用深度学习完成肝脏CT图像分割任务时采用的多为全监督的方式. 全监督深度学习方法的性能在较大程度上依赖于标注数据的数量和质量[22]. 然而医学图像的标注耗时耗力. 据统计,手动分割一个肝脏序列平均需要30 min以上[23],且对专家经验依赖性较强,因此获得大量高质量的CT图像肝脏标注数据非常困难. 半监督方法被广泛地应用到了深度学习图像分割领域. 近几年,研究者们提出了多种基于半监督学习的图像分割方法,主要包括3种类型[24]:自训练[25-26]、协同训练[27]、基于图的方法[28]. Zhou等[29]提出多平面联合训练的方法,将三维腹部CT图像用矢状向、冠状向及轴向3个平面表示,分别以逐片方式和少量标注进行训练. Jiang等[30]将图学习操作和传统图卷积网络结构集成在一个网络中,学习最适合半监督图卷积网络的最优图结构. 然而,协同训练使用三维CT图像的切片进行训练[31],不能充分利用图像的层间信息;基于图的半监督学习须假设图的标签是平滑的,有一定的限制条件. 由于自训练学习方法简单有效,不需要特定的假设条件[32],在许多计算机视觉问题上取得出色的成绩,在实际中应用广泛. 本研究将基于自训练的半监督学习方法用于3D scSE-UNet的训练,应用于三维肝脏CT图像的分割任务中,以缓解大量高质量标注图像难以获得的问题.
本研究创新点在于:1)提出基于自训练3D scSE-UNet分割网络的半监督学习肝脏分割方法. 所提出的3D scSE-UNet的特性在于,在3D UNet中引入了改进的scSE-block+. 相比于经典的scSE-block,scSE-block+包含全局最大池化层(global max pooling,GMP),可以更好地保留图像边缘信息,其特征学习能力也得到进一步提升,有利于提升网络的分割精度. 2)在自训练过程中加入全连接条件随机场[33](dense conditional random field,Dense CRF),对预测得到的伪标签进行边缘细化处理,提高半监督分割网络产生的伪标签的精确度. 本研究使用LiTS17 Challenge数据集和SLIVER07数据集进行肝脏分割实验. 该方法实现了联合使用少量标记数据和大量未标记数据对3D scSE-UNet进行有效训练,从而提升肝脏分割精度的目的,降低了深度学习图像分割方法对标签数据的依赖性.
1. 基于3D scSE-UNet的半监督深度学习自动分割方法
1.1. 自训练半监督学习分割框架
为了充分利用影像中的有效信息,提升目标区域的自动分割精确度,采用自训练方式进行分割. 向分割网络同时输入少量有标签数据和大量无标签数据实现网络的循环迭代训练.
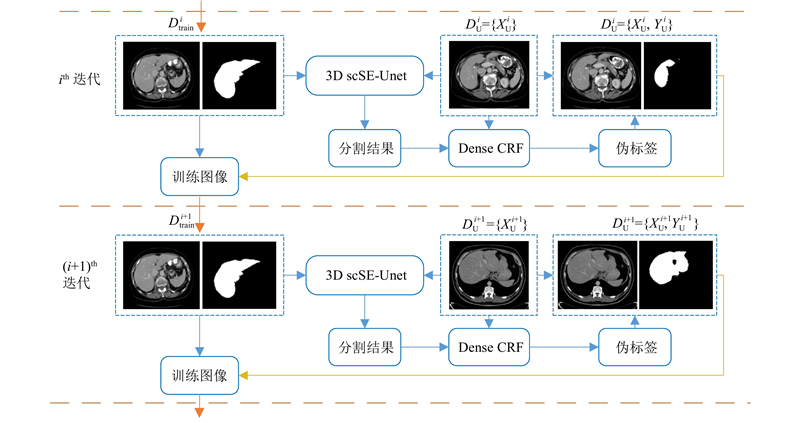
本研究的自训练半监督分割方法如图1所示. 图中,X为灰度图像,Y为其对应的标签图像. 该方法使用2个数据集,一个是带标签图像的数据集
图 1
图 1 基于3D scSE-UNet的自训练半监督方法示意图
Fig.1 Illustration of self-training semi-supervised method based on 3D scSE-UNet
具体训练步骤如下:1)用带标签图像的数据集DL训练3D scSE-UNet分割网络,即
1.2. 3D scSE-UNet 分割网络
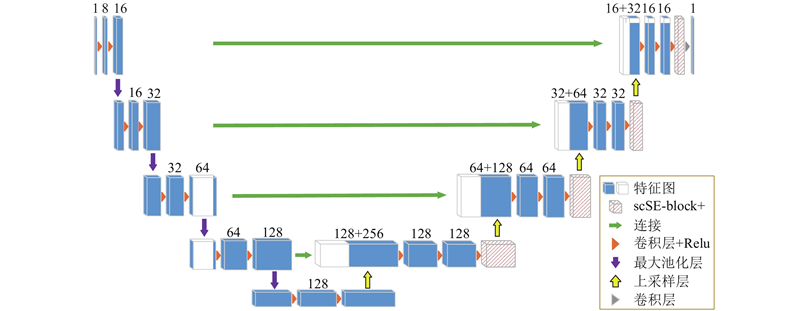
在所提出的半监督学习分割方法中,将scSE-block+作为附加层与3D UNet相结合,构成3D scSE-UNet分割网络,其网络结构如图2所示. 其整体框架与3D UNet的结构类似,同样采用U型结构,包含编码部分和解码部分. 图中,蓝色方块为训练过程中产生的特征图,方块上方的数字表示该特征图的通道数.
图 2
在3D UNet解码部分的每一个跳跃连接层的末端分别添加一个scSE-block+,如图2中红色斜线方块所示. 该模块重新标定了特征通道权重,强化有用特征通道,抑制无关特征通道,从而提升分割网络自动学习有效特征的能力.
所提出的3D scSE-UNet模型的具体结构如表1所示. 编码部分通过下采样分析输入图像并进行特征提取;解码部分通过上采样生成与原始图像相同大小的特征图. 在编码路径中包含4个下采样层,在每个下采样层中由2个卷积层提取不同层次的图像特征,并使用ReLU函数进行激活,同时紧接一个步长为2的最大池化层压缩特征,减少参数量. 在解码路径中,每一层包含一个步长为2的上采样层,再紧跟2个3×3×3的卷积层,每一个卷积层后面依然接一个ReLU层. 通过跳跃连接,将编码阶段获得的特征图同解码阶段获得的分辨率相同的特征图融合在一起,结合浅层次和深层次的特征细化图像,允许更多的原图像纹理信息在高分辨率的层中进行传播. 将跳跃连接融合后的特征输入scSE-block+以抑制不重要特征,提高分割结果的准确度. 最后一层为1×1×1的卷积层,输出通道数为标签类别数量的预测分割图.
表 1 3D scSE-UNet网络结构表
Tab.1
| 网络结构层 1) | 特征图大小 | 卷积核参数 | 网络结构层 | 特征图大小 | 卷积核参数 | |
| 1)注:第2列表示当前层的输出特征的大小及通道数; 第3列中[ ]表示卷积操作,“3×3×3,8”表示经过卷积核大小为3×3×3和8通道的卷积层; “Dropout_1+UpSampling3D_1”中“+”表示Concatenate_1是将Dropout_1与UpSampling3D_1跳跃连接. | ||||||
| input | 128×128×64×1 | − | scSE_block_1 | 16×16×8×128 | − | |
| Conv3D_1 | | | UpSampling3D_2 | 32×32×16×128 | 2×2×2 | |
| MaxPooling3D_1 | 64×64×32×16 | 2×2×2 | Concatenate_2 | 32×32×16×192 | Conv3D_3+UpSampling3D_2 | |
| Conv3D_2 | | | Conv3D_7 | | | |
| MaxPooling3D_2 | 32×32×16×32 | 2×2×2 | scSE_block_2 | 32×32×16×64 | − | |
| Conv3D_3 | | | UpSampling3D_3 | 64×64×32×64 | 2×2×2 | |
| MaxPooling3D_3 | 16×16×8×64 | 2×2×2 | Concatenate_3 | 64×64×32×96 | Conv3D_2+UpSampling3D_3 | |
| Conv3D_4 | | | Conv3D_8 | 64×64×32×32 | | |
| Dropout_1 | 16×16×8×128 | 0.5 | scSE_block_3 | 64×64×32×32 | − | |
| MaxPooling3D_4 | 8×8×4×128 | 2×2×2 | UpSampling3D_4 | 128×128×64×32 | 2×2×2 | |
| Conv3D_5 | | | Concatenate_4 | 128×128×64×48 | Conv3D_1+UpSampling3D_4 | |
| Dropout_2 | 8×8×4×256 | 0.5 | Conv3D_9 | 128×128×64×16 | | |
| UpSampling3D_1 | 16×16×8×256 | 2×2×2 | scSE_block_4 | 128×128×64×16 | − | |
| Concatenate_1 | 16×16×8×384 | Dropout_1+UpSampling3D_1 | Conv3D_10 | 128×128×64×1 | [1×1×1,1] | |
| Conv3D_6 | | | − | − | − | |
1.3. scSE-block+模块
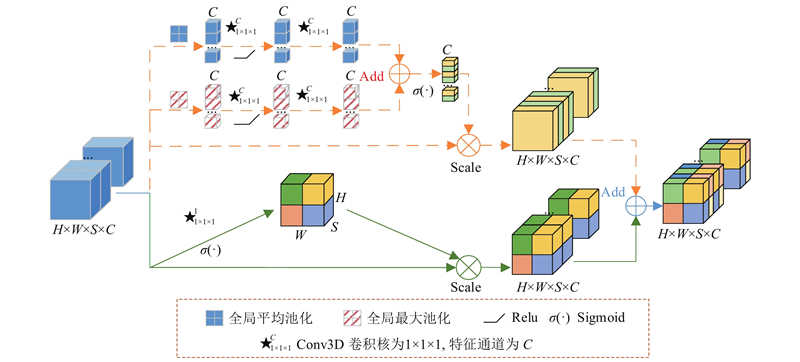
在3D scSE-UNet中使用4个scSE-block+模块,其细节结构如图3所示. 该模块可以同时关注特征空间域和特征通道域,通过自动学习每个特征的重要程度重新标定特征图. scSE-block+是通道SE模块(channel SE-block,cSE-block)和空间SE模块(spatial SE-block,sSE-block)的加和.
图 3
如图3所示,对cSE-block进行改进,在图中表示为虚线箭头部分. 在cSE-block的基础上加入全局最大池化层,在图中表示为红色斜线方块,该层与cSE-block原有的全局平均池化层并行. 在压缩空间信息、将图像空间特征张量压缩成向量的时候,全局最大池化层和全局平均池化层分别会选择不同的空间特征张量进行压缩.
全局最大池化层对整张特征图取最大值,由于图像边缘可能会产生最大的特征值,全局最大池化层可以较好地保留图像纹理、边缘特征. 全局平均池化层是对整张特征图取平均值,强调对整体特征进行下采样,可以较好地保留背景. 因此对于图像边缘信息的提取,采用全局最大池化层会比采用全局平均池化层更有效. 并行使用这2个层可以保留边缘和背景2路特征,在提高模型性能方面的作用明显. 具体地,cSE-block通过压缩空间信息来衡量通道的重要性,仅沿通道方向激发. 模块将H×W×S×C大小的特征图并行通过一个全局平均池化层和一个全局最大池化层,其中,H、W、S、C分别表示特征图的长度、宽度、深度和通道数. 这2个池化层分别将每个通道上的全局空间信息压缩为1个张量数值,分别产生1×C个特征值. 然后分别对其进行卷积核为1×1×1、通道数为C的三维卷积操作,将卷积结果分别通过非线性激活函数ReLU,将所得到的输出再分别经过相同的卷积操作,最终得到2个数值不同但维度相同的1×1×1×C的张量. 将这2个张量相加之后通过sigmoid层将每个值归一化到值域[0,1.0],与原本特征矩阵相乘后能明显抑制不重要通道内的信息,保持重要通道内的信息几乎不变,变相提升有效特征的提取.
最后scSE-block+可以分别沿通道和空间重新校准特征图,然后合并输出,从而有效利用特征图在空间和通道2方面的有效信息.
1.4. 全连接条件随机场
在自训练中,通过3D scSE-UNet模型分割无标记样本时容易产生错误. 分割模型产生错误的预测结果后,仍会将其作为该样本的标记一起扩充至训练集进行下一轮的训练,在一定程度上会影响后续训练过程中训练数据的质量,从而降低下一次迭代训练的效果,最终造成错误累积,甚至将错误放大.
为了降低错误累积的影响,须在最大程度上提高伪标签的精确度. 全连接条件随机场可以优化伪标签中粗糙和不确定的标记,修正细碎的错分区域,改善网络的定位特性,进而得到更精确细致的伪标签. 因此,本研究在自训练过程中引入Dense CRF,对自训练过程中每一次迭代得到的伪标签进行细化处理.
Dense CRF描述的是像素点与像素点之间的关系,鼓励相似像素分配相同的标签,即被分割的可能性小;而相似度较低的像素分配不同标签,即被分割的可能性大. Dense CRF的能量函数[33]如下:
式中:
式中:ω1、ω2为线性组合权重; µ为标签兼容性函数,
具体地,在本研究使用Dense CRF处理分割结果的过程中,一元势能为概率分布图,即由模型输出的特征图经过softmax函数运算得到的结果;二元势能中的位置信息和灰度信息由原始图像提供. 将一元势能与二元势能结合起来可以较全面地考量像素之间的关系,并得出优化后的结果. 本研究通过迭代能量函数E(x),每个图像通过5次迭代寻找最小解,找出该图像中每个体素最可能归属的类别,从而优化预测结果. 最终,将每个优化处理后的分割结果作为伪标签加入自训练的迭代中.
通过Dense CRF对分割结果的优化,提高伪标签的准确度,从而减小伪标签与真实值之间的误差,避免由于错误累积过大导致分割模型性能降低.
2. 数据及模型
2.1. 数据及预处理
本研究实验数据为来自LiTS17 Challenge数据集和SLIVER07数据集中训练数据的肝脏CT图像. 将获得的数据集进行整理,最终得到146例样本的肝CT图像及其对应的医生分割标签,用于接下来的分割处理. 肝CT图像大小为512×512×L(其中L为CT数据中的切片数量,范围为32~375),切片内X、Y轴方向上的分辨率为0.56~1.00 mm,切片间距为0.7~5.0 mm.
在不同病人间,肝脏CT图像的灰度分布范围存在一定的差异,为了降低灰度分布范围的差异性对脏器自动分割结果的影响,将肝脏CT图像的灰度值标准化到[−100,300]. 同时,对数据进行旋转变换,使数据量增加至2倍,提高网络在有限数据情况下的训练精度.
2.2. 模型训练
在实验设置方面,将1.1节步骤2)中的n设置为5,将无标签图像的数据集随机分为5个子数据集依次输入分割模型中进行预测,产生对应的分割结果. 在训练过程中使用Adam优化器实现梯度下降算法,寻找使误差函数最小的网络参数. 设置网络学习率为0.0001;设置epoch=150,到最大epoch数时停止训练;设置batch size=1. 所有模型的训练、验证及测试均在同一台计算机上运行. 其配置如下:Intel i7-9700k@3.60 GHz CPU和NVIDIA RTX 2080Ti显卡. 主要的软件环境:Python 3.6、CUDA 10.1、Keras2.2.5等.
2.3. 评价指标
对于分割任务,通过计算模型自动分割图像和专家手动分割图像之间的Dice相似系数(dice similarity coefficient,DSC)来评估分割性能. 体素重叠误差(volume overlap error,VOE)与DSC是相同评估指标的不同表述. 相对体积差(relative volume difference,RVD)[35]表示两者体积之间的差异. VOE和RVD用百分比表示. 上述指标的计算公式如下:
式中:P为预测得到的目标区域,G为医生勾画的目标区域.
敏感度(sensitivity,SEN)能有效反映分割结果对目标区域的敏感程度. 阳性预测值(positive predicted value,PPV)能得到真实目标区域在检测出的阳性区域中所占比例. 敏感度和阳性预测值的表达式如下:
式中:TP为真实阳性,即被正确分为目标区域的像素个数;TN为真实阴性,即被正确分为背景区域的像素个数;FP为假阳性,即被错误分为目标区域的像素个数;FN为假阴性,即被错误分为背景区域的像素个数.
平均对称表面距离(average symmetric surface distance,ASD)为P中每个表面体素与G中最近的表面体素之间距离的平均值. 对称位置表面距离的均方根(root mean square symmetric surface distance,RMSD)也是距离测度的评价标准. 最大对称表面距离(maximum symmetric surface distance,MSD)为P的表面体素和G的表面体素之间的最大差异. 上述3个距离指标均以毫米为单位,表达式如下:
式中:S(P)、S(G)分别为P、G表面体素的集合.
3. 实验结果与分析
3.1. 不同标签占比的结果
基于本研究提出的半监督分割方法进行5次实验,为了避免实验结果的偶然性,所有实验结果均取5次实验的均值作为最终结果. 本研究实验将全监督3D UNet分割方法作为基线方法.
每次实验将SLIVER07数据集中训练数据的20例图像,以及在126例LiTS17 Challenge数据集中随机选取的26例图像,共46例肝脏CT图像作为测试集. 在LiTS17 Challenge数据集剩余的100例数据中,随机选取50、40、30、20、10例灰度图像及其真值标注作为有标签数据,其余50、60、70、80、90例灰度图像作为无标签数据,构成了不同有标签数据占比下的训练集. 将这几种实验设置下获得的模型分别对测试集进行分割预测,将其结果与3D UNet使用100例灰度图像及其真值标注的全监督分割结果进行对比. 基线方法与本研究的半监督方法对肝脏分割表现出了不同的性能,得到的结果如表2所示. 表中,L为训练集中有标签的图像数量,U为训练集中无标签的图像数量,LP为训练集中有标签的图像占训练集所有图像的比例.可以看出,当肝脏分割中训练集有标签数据的占比LP=30%时,本研究方法分割DSC=0.941,SEN=0.959,PPV=0.925;而基线方法使用100个有标签数据训练得到的分割DSC=0.937,SEN=0.958,PPV=0.920. 由此可得,在该占比下本研究半监督分割方法的DSC已经可以超过基线方法的DSC,距离指标也均优于基线方法.
表 2 不同标签数量占比下的分割性能比较
Tab.2
| 方法 | L | U | LP/% | DSC | SEN | PPV | VOE/% | RVD/% | ASD/mm | RMSD/mm | MSD/mm |
| 全监督3D UNet | 100 | − | − | 0.937±0.034 | 0.958±0.021 | 0.920±0.059 | 5.613±6.744 | 6.034±7.788 | 4.168±5.688 | 10.133±13.870 | 88.078±61.122 |
| 全监督3D scSE-UNet | 100 | − | − | 0.950±0.032 | 0.964±0.047 | 0.938±0.041 | 4.532±6.572 | 4.689±6.443 | 1.974±2.032 | 4.257±4.660 | 38.767±22.557 |
| 半监督3D scSE-UNet | 50 | 50 | 50 | 0.948±0.023 | 0.960±0.024 | 0.937±0.041 | 4.162±4.696 | 4.262±5.078 | 2.037±1.123 | 4.227±2.571 | 37.608±16.748 |
| 40 | 60 | 40 | 0.946±0.025 | 0.959±0.026 | 0.934±0.048 | 5.079±5.358 | 4.970±6.004 | 2.143±1.213 | 4.295±2.801 | 36.659±16.682 | |
| 30 | 70 | 30 | 0.941±0.032 | 0.959±0.047 | 0.925±0.045 | 5.130±6.832 | 5.176±6.853 | 2.431±1.702 | 5.014±3.798 | 40.554±19.559 | |
| 20 | 80 | 20 | 0.930±0.040 | 0.945±0.048 | 0.919±0.060 | 6.326±8.065 | 6.598±8.684 | 2.972±2.176 | 5.758±4.541 | 43.552±21.964 | |
| 10 | 90 | 10 | 0.902±0.049 | 0.943±0.052 | 0.874±0.078 | 11.337±10.201 | 12.419±12.079 | 4.403±2.736 | 8.000±4.958 | 49.131±21.866 |
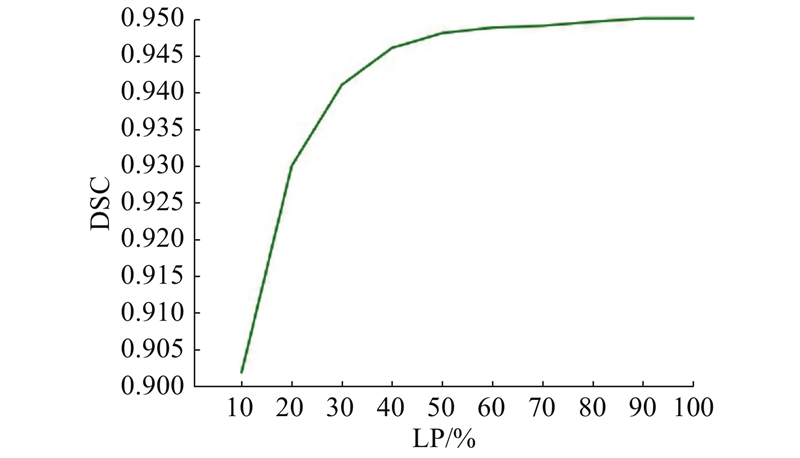
在本研究提出的3D scSE-UNet分割网络基础上,对训练集不同标签数量占比下该网络的分割性能进行比较,得到dice score曲线如图4所示. 在标签数量占比到40%之前,随着标签数量占比的不断增加,3D scSE-UNet模型分割结果的DSC提升较快,从0.902快速提升到0.946;在40%之后DSC提升缓慢;直至标签占比达到100%,即全监督时3D scSE-UNet模型分割结果的DSC=0.950.
图 4
图 4 不同标签数量占比下的3D scSE-UNet分割dice score曲线
Fig.4 Dice score curve of 3D scSE-UNet with different proportions of labels
为了能够直观展示本研究方法的分割结果,在有标签占比为30%的分割结果中,随机选取4例CT图像,如图5所示. 将真值标注、基线方法得到的分割结果及本研究的半监督3D scSE-UNet方法得到的分割结果进行直观比较.
图 5
图 5 基线方法分割结果与本研究半监督3D scSE-UNet方法分割结果对比
Fig.5 Comparison of segmentation results between baseline and semi-supervised 3D scSE-UNet methods
本研究提出的3D scSE-UNet半监督框架可以达到与基线方法全监督3D UNet相当的分割结果,也可以达到与真值标注相近的结果. 因此该自训练3D scSE-UNet半监督框架可以在一定程度上代替全监督3D UNet框架.
3.2. 半监督学习与全监督性能比较
为了验证本研究使用的自训练半监督框架的有效性,在本研究提出的3D scSE-UNet分割网络的基础上,对不同占比下的有标签数据进行全监督和半监督的分割性能比较,得到结果如表3所示. 可以看出,在同时使用3D scSE-UNet分割网络,并使用相同数量有标签数据的情况下,半监督学习可以通过对额外的无标签数据的有效利用,进一步提高分割性能,这在有标签数据较少的情况下尤为明显. 在有标签数据较多的情况下,半监督分割精度提升不明显. 当有标签数量占比为50%时,全监督和半监督的DSC相差较小,但仍未持平. 这可能是由于当有较多的有标签数据可用时,分割网络已经可以训练得较好,半监督学习带来的改进也会变小.
表 3 不同标签数量占比下使用3D scSE-UNet进行全监督和半监督的分割性能比较
Tab.3
| 监督方式 | L | U | LP/% | DSC | SEN | PPV | VOE/% | RVD/% | ASD/mm | RMSD/mm | MSD/mm |
| 全监督 | 10 | − | − | 0.896±0.053 | 0.934±0.054 | 0.867±0.082 | 11.283±10.339 | 12.333±12.343 | 9.203±7.892 | 21.924±17.558 | 140.408±63.219 |
| 半监督 | 10 | 90 | 10 | 0.902±0.049 | 0.943±0.052 | 0.874±0.078 | 11.337±10.201 | 12.419±12.079 | 4.403±2.736 | 8.000±4.958 | 49.131±21.866 |
| 全监督 | 20 | − | − | 0.914±0.065 | 0.941±0.060 | 0.893±0.086 | 7.860±10.219 | 8.721±12.834 | 5.882±8.069 | 13.767±15.446 | 115.778±69.480 |
| 半监督 | 20 | 80 | 20 | 0.930±0.040 | 0.945±0.048 | 0.919±0.060 | 6.326±8.065 | 6.598±8.684 | 2.972±2.176 | 5.758±4.541 | 43.552±21.964 |
| 全监督 | 30 | − | − | 0.925±0.045 | 0.953±0.057 | 0.903±0.067 | 7.786±9.618 | 8.356±10.889 | 4.301±2.755 | 10.548±5.728 | 81.865±25.524 |
| 半监督 | 30 | 70 | 30 | 0.941±0.032 | 0.959±0.047 | 0.925±0.045 | 5.130±6.832 | 5.176±6.853 | 2.431±1.702 | 5.014±3.798 | 40.554±19.559 |
| 全监督 | 40 | − | − | 0.937±0.039 | 0.950±0.036 | 0.928±0.064 | 5.549±7.023 | 5.847±7.830 | 3.821±3.949 | 6.258±10.666 | 59.050±67.107 |
| 半监督 | 40 | 60 | 40 | 0.946±0.025 | 0.959±0.026 | 0.934±0.048 | 5.079±5.358 | 4.970±6.004 | 2.143±1.213 | 4.295±2.801 | 36.659±16.682 |
| 全监督 | 50 | − | − | 0.943±0.026 | 0.960±0.028 | 0.929±0.044 | 4.717±5.280 | 4.912±5.702 | 2.230±1.212 | 4.452±2.651 | 36.582±14.887 |
| 半监督 | 50 | 50 | 50 | 0.948±0.023 | 0.960±0.024 | 0.937±0.041 | 4.162±4.696 | 4.262±5.078 | 2.037±1.123 | 4.227±2.571 | 37.608±16.748 |
3.3. 融合scSE-block+的分割网络对比
为了验证本研究提出的scSE-block+这一模块对网络分割性能的提升能力,在自训练的半监督框架下,对3D UNet和3D scSE-UNet的分割结果进行比较. 实验设置在有标签数据占比为30%的条件下,结果如表4所示. 可以看出,将3D UNet与本研究提出的scSE-block+相结合后的分割网络的性能优于3D UNet,除了DSC明显提升外,SEN和PPV也都有所提升,体积误差明显降低,距离指标也优于3D UNet. 可见scSE-block+激发了特征图在空间和通道上的更多信息,充分挖掘了图像高级语义信息. 因此本研究提出的3D scSE-UNet网络模型可以有效提高肝脏分割精度.
表 4 3D UNet与3D scSE-UNet性能对比
Tab.4
| 方法 | DSC | SEN | PPV | VOE/% | RVD/% | ASD/mm | RMSD/mm | MSD/mm |
| 半监督3D UNet | 0.935±0.032 | 0.955±0.045 | 0.921±0.049 | 6.458±7.041 | 6.740±7.681 | 3.493±1.596 | 8.476±3.617 | 66.090±19.594 |
| 半监督3D scSE-UNet | 0.941±0.032 | 0.959±0.047 | 0.925±0.045 | 5.130±6.832 | 5.176±6.853 | 2.431±1.702 | 5.014±3.798 | 40.554±19.559 |
3.4. scSE-block+改进前、后性能比较
为了验证在scSE-block中加入全局最大池化层构成的scSE-block+的有效性,评估该模块改进前、后对半监督分割结果的影响. 实验仍然是在有标签数据占比为30%的情况下完成的,结果如表5所示. 可以看出,在原scSE-block基础上加入全局最大池化后,可以得到比原scSE-block更好的效果. 在特征提取中利用全局最大池化层可以更多地保留图像边缘信息,优化分割网络对目标区域的分割效果.
表 5 scSE-block+改进前、后性能对比
Tab.5
| 方法 | DSC | SEN | PPV | VOE/% | RVD/% | ASD/mm | RMSD/mm | MSD/mm |
| scSE-block | 0.939±0.030 | 0.951±0.044 | 0.928±0.049 | 5.464±6.775 | 5.583±7.124 | 2.535±1.693 | 5.162±3.824 | 40.689±18.774 |
| scSE-block+ | 0.941±0.032 | 0.959±0.047 | 0.925±0.045 | 5.130±6.832 | 5.176±6.853 | 2.431±1.702 | 5.014±3.798 | 40.554±19.559 |
3.5. Dense CRF性能比较
为了验证在自训练过程中加入Dense CRF的有效性,对伪标签细化处理前、后的分割效果进行对比,如表6所示,该实验依然在有标签数据占比为30%的条件下进行.
表 6 Dense CRF处理前、后分割性能对比
Tab.6
| Dense CRF | DSC | SEN | PPV | VOE/% | RVD/% | ASD/mm | RMSD/mm | MSD/mm |
| 不使用 | 0.940±0.036 | 0.956±0.045 | 0.922±0.047 | 4.544±6.371 | 4.625±6.580 | 2.417±3.606 | 5.097±10.050 | 48.937±.32.456 |
| 使用 | 0.941±0.032 | 0.959±0.047 | 0.925±0.045 | 5.130±6.832 | 5.176±6.853 | 2.431±1.702 | 5.014±3.798 | 40.554±19.559 |
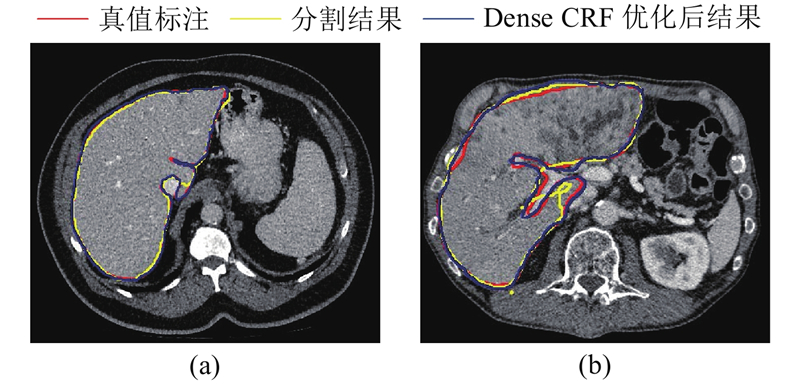
图 6
图 6 Dense CRF优化后的结果与真值标注、网络分割结果的对比
Fig.6 Comparison of optimized result of Dense CRF with ground truth and result of network segmentation
Dense CRF对分割网络预测的概率图进行精细处理,进一步考虑单个像素和其他所有像素的关系,在图像中的所有像素对上建立依赖关系. 在半监督自训练框架中加入Dense CRF,尽管对于整体分割精度的提升作用有限,但对于分割边缘的细化有较大帮助.
3.6. 模型时间性能比较
对本研究对比实验中的不同算法模型进行时间性能的比较. 使用4种不同的模型,即本研究半监督3D scSE-UNet方法的分割模型、基线方法全监督3D UNet的分割模型、全监督3D scSE-UNet方法的分割模型、半监督3D UNet方法的分割模型. 在同一例三维肝脏CT图像上自动分割出肝脏区域的时间如表7所示. 表中, T为分割模型对单例三维图像进行预测的时间. 该例三维肝脏CT图像有109张切片.可以看出,由于scSE-block+的加入,虽然3D scSE-UNet分割模型处理图像的时间比3D UNet分割模型处理图像的时间长,但两者相差在150 ms以内,只是在较小程度上增加了计算时间,在临床使用可接受的范围内. 对于同一种分割网络,使用半监督和全监督框架的分割模型处理图像的时间近似,说明监督方式对于模型的处理时间影响较小,可以忽略不计.
表 7 不同模型平均处理图像时间
Tab.7
| 模型 | T/ms |
| 全监督3D UNet | 747 |
| 半监督3D UNet | 769 |
| 全监督3D scSE-UNet | 895 |
| 半监督3D scSE-UNet | 893 |
4. 结 语
针对基于深度学习的医学图像分割需要大量标注数据、专家标注费时的问题,提出基于scSE-block+的3D scSE-UNet肝脏CT图像半监督学习分割方法,有效降低了深度学习方法在医学图像分割任务中对有标签数据的依赖. 其中scSE-block+是在scSE-block的通道注意力上加入一条新的并行的基于全局最大池化的通道注意力,更好地保留图像纹理边缘信息,提升有用特征并抑制无关特征;半监督学习部分是把未标注的数据随机分成n份,依次进行监督学习、预测其中一份未标注数据、将预测结果经过Dense CRF调整优化后作为伪标签参与下一轮训练,直到用完所有未标注数据. 实验结果表明全连接条件随机场可以对分割结果进行细化处理,提高分割网络产生伪标签的精确度;半监督学习中伪标签的加入能够提升模型的分割性能,并可以达到用少量标注实现大量预测的效果. 将本研究的方法在肝脏CT图像数据集(LiTS17 Challenge、SLIVER07)中进行验证,测试结果表明仅使用少量的有标签数据可以达到与全监督分割结果相当的水平.
本研究所提出的3D scSE-UNet模型通过显式地学习不同特征之间的联系,提升有用特征并抑制无关特征,从而提高肝脏区域的分割精度。但该模型由于scSE-block+的引入,不论是哪种监督方式,3D scSE-UNet模型处理图像的时间均比3D UNet模型平均增加17.941%,相差在150 ms以内;3D scSE-UNet模型的参数也更多,比3D UNet模型增加了2.143%。但是就处理单例三维图像而言,3D scSE-UNet模型的处理时间仍在临床使用可接受范围内。并且在相同的监督方式下,3D scSE-UNet模型的分割性能明显优于3D UNet模型。因此,认为3D scSE-UNet模型是有意义和应用价值的。仍存在以下问题须在今后的工作中进一步探讨。本研究使用的自训练半监督方法如果在未标记数据的初始分割中出现错误或偏差(分割过度或分割不足),网络将在随后的迭代训练中学习错误,从而在一次次迭代中将该错误放大。这种负面影响目前通过在自训练过程中使用Dense CRF优化分割结果来缓解,并假设大多数自动分割是正确的,因此网络学习的平均梯度仍然大致正确[33]。在之后的工作中,将尝试把分割结果不确定性估计融入半监督学习中。还会将本研究提出的半监督分割方法应用到其他医学图像数据集中,以提高该方法的普适性。
参考文献
Computer-aided surgery meets predictive, preventive and personalized medicine
[J].DOI:10.1007/s13167-017-0084-8 [本文引用: 1]
Automated lung seg- mentation and smoothing techniques for inclusion of juxtapleural nodules and pulmonary vessels on chest CT images
[J].DOI:10.1016/j.bspc.2014.03.010 [本文引用: 1]
Low-rank and sparse decomposition based shape model and probabilistic atlas for automatic pathological organ segmentation
[J].DOI:10.1016/j.media.2017.02.008 [本文引用: 1]
A hierarchical local region-based sparse shape composition for liver segmentation in CT scans
[J].DOI:10.1016/j.patcog.2015.09.001 [本文引用: 1]
Automatic liver segmentation based on shape constraints and deformable graph cut in CT images
[J].DOI:10.1109/TIP.2015.2481326 [本文引用: 1]
Liver CT sequence segmentation based with improved U-Net and graph cut
[J].DOI:10.1016/j.eswa.2019.01.055 [本文引用: 1]
The study and application of the improved region growing algorithm for liver segmentation
[J].DOI:10.1016/j.ijleo.2013.10.049 [本文引用: 1]
基于区域增长与统一化水平集的CT肝脏图像分割
[J].
Liver segmentation in CT images based on region-growing and unified level set method
[J].
Shape-intensity prior level set: combining probabilistic atlas and probability map constrains for automatic liver segmentation from abdominal CT images
[J].DOI:10.1007/s11548-015-1332-9 [本文引用: 1]
H-DenseUNet: hybrid densely connected UNet for liver and tumor segmentation from CT volumes
[J].DOI:10.1109/TMI.2018.2845918 [本文引用: 1]
Fully convolutional networks for semantic segmentation
[J].
基于新型深度全卷积网络的肝脏CT影像三维区域自动分割
[J].DOI:10.3969/j.issn.0258-8021.2018.04.001 [本文引用: 1]
A new fully convolutional network for 3D liver region segmentation on CT images
[J].DOI:10.3969/j.issn.0258-8021.2018.04.001 [本文引用: 1]
结合改进的U-Net和Morphsnakes的肝脏分割
[J].
Liver segmentation with improved U-Net and Morphsnakes algorithm
[J].
Multi-scale attention module U-Net liver tumour segmentation method
[J].DOI:10.1088/1742-6596/1678/1/012107 [本文引用: 1]
融合零样本学习和小样本学习的弱监督学习方法综述
[J].DOI:10.3969/j.issn.1001-506X.2020.10.13 [本文引用: 1]
Survey of weakly supervised learning integrating zero-shot and few-shot learing
[J].DOI:10.3969/j.issn.1001-506X.2020.10.13 [本文引用: 1]
Automated hepatic volumetry for living related liver transplantation at multisection CT
[J].DOI:10.1148/radiol.2403050850 [本文引用: 1]
Not-so- supervised: a survey of semi-supervised, multi-instance, and transfer learning in medical image analysis
[J].DOI:10.1016/j.media.2019.03.009 [本文引用: 1]
Semi-supervised cerebrovascular segmentation by hierarchical convolutional neural network
[J].DOI:10.1109/ACCESS.2018.2879521 [本文引用: 1]
基于深度协同训练的肝脏CT图像自动分割方法
[J].
Automatic liver segmentation from CT images based on deep co-training
[J].
Efficient inference in fully connected CRFs with Gaussian edge potentials
[J].