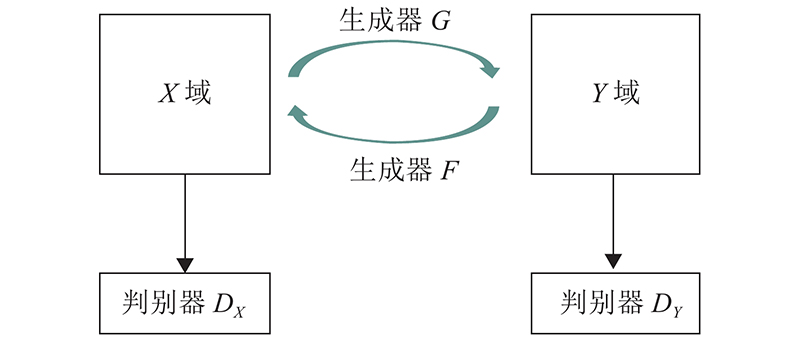
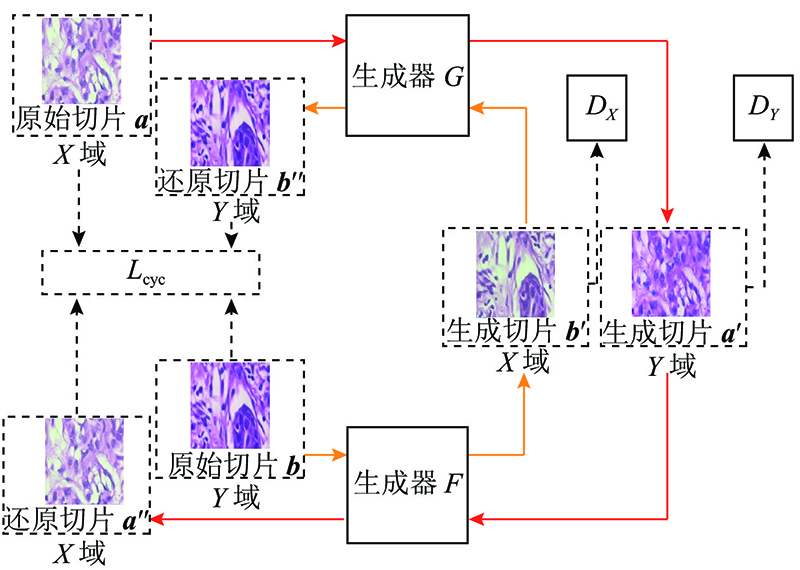
Cycle-GAN总体训练的损失函数由以下3部分组成. 1)X 域GAN的损失: ${L_{{\rm{GAN}}}}\left( {F,{D_X},Y,X} \right)$ ${{D_X}}$ $F$ . 2)Y 域GAN的损失: ${L_{{\rm{GAN}}}}\left( {G,{D_Y},X,Y} \right)$ ${D_Y}$ $G$ . 3)重构误差: ${L_{{\rm{cyc}}}} = L_{{\rm{cyc}}}^x + L_{{\rm{cyc}}}^y$ .
(1) $ \begin{split} L(G,F,{{D_X}},{{D_Y}}) =& {L_{{\rm{GAN}}}}\left( {G,{D_Y},X,Y} \right) + \\ & {L_{{\rm{GAN}}}}\left( {F,{D_X},Y,X} \right) + \lambda {L_{{\rm{cyc}}}}(G,F). \end{split} $
[1]
CHEN W, ZHENG R, BAADE P, et al Cancer Statistics in China, 2015
[J]. CA: A Cancer Journal for Clinicians , 2016 , 66 (2 ): 115 - 132
DOI:10.3322/caac.21338
[本文引用: 1]
[2]
KOWAL M, FILIPCZUK P, OBUCHOWICZ A, et al Computer-aided diagnosis of breast cancer based on fine needle biopsy microscopic images
[J]. Computers in Biology and Medicine , 2013 , 43 (10 ): 1563 - 1572
DOI:10.1016/j.compbiomed.2013.08.003
[本文引用: 1]
[3]
ISABEL G, POLONIA A, SARMIENTO A, et al Automatic classification of tissue malignancy for breast carcinoma diagnosis
[J]. Computers in Biology and Medicine , 2018 , 96 : 41 - 51
DOI:10.1016/j.compbiomed.2018.03.003
[本文引用: 2]
[4]
ABDULLAH-AL N, YINAN K Histopathological breast-image classification using concatenated R-G-B histogram information
[J]. Annals of Data Science , 2019 , 6 (3 ): 513 - 529
DOI:10.1007/s40745-018-0162-3
[本文引用: 2]
[5]
WANG P, HU X, LI Y, et al Automatic cell nuclei segmentation and classification of breast cancer histopathology images
[J]. Signal Processing , 2016 , 122 : 1 - 13
DOI:10.1016/j.sigpro.2015.11.011
[本文引用: 1]
[6]
SPANHOL F, OLIVEIRA L, PETITJEAN C, et al A dataset for breast cancer histopathological image classification
[J]. IEEE Transactions on Biomedical Engineering , 2016 , 63 (7 ): 1455 - 1462
DOI:10.1109/TBME.2015.2496264
[本文引用: 1]
[7]
VETA M, PLUIM J, VAN D, et al Breast cancer histopathology image analysis: a rcview
[J]. IEEE Transactions on Biomedical Engineering , 2014 , 61 (5 ): 1400 - 1411
DOI:10.1109/TBME.2014.2303852
[本文引用: 1]
[9]
SPANHOL F, OLIVEIRA L, PETITJEAN C, et al. Breast cancer histopathological image classification using convolutional neural networks [C]// Proceedings of 2016 International Joint Conference on Neural Networks. Vancouver: IEEE, 2016: 2560-2567.
[本文引用: 2]
[10]
BAYRAMOGLU N, KANNALA J, JANNE H. Deep learning for magnification independent breast cancer histopathology image classification [C]// Proceedings of International Conference on Pattern Recognition . Cancun: IEEE, 2016: 2441-2446.
[本文引用: 2]
[12]
明涛, 王丹, 郭继昌, 等 基于多尺度通道重校准的乳腺癌病理图像分类
[J]. 浙江大学学报:工学版 , 2020 , 54 (7 ): 1289 - 1297
[本文引用: 2]
MING Tao, WANG Dan, GUO Ji-chang, et al Breast cancer histopathological image classification using multi-scale channel squeeze-and-excitation model
[J]. Journal of Zhejiang University: Engineering Science , 2020 , 54 (7 ): 1289 - 1297
[本文引用: 2]
[13]
邹文凯, 陆慧娟, 叶敏超, 等 基于卷积神经网络的乳腺癌组织病理图像分类
[J]. 计算机工程与设计 , 2020 , 41 (6 ): 1749 - 1754
[本文引用: 2]
ZOU Wen-kai, LU Hui-juan, YE Min-chao, et al Breast cancer histopathological image classification using convolutional neural network
[J]. Computer Engineering and Design , 2020 , 41 (6 ): 1749 - 1754
[本文引用: 2]
[14]
NAHID A, MEHRABI M, KONG Y. Frequency-domain information along with LSTM and GRU methods for histopathological breast-image classification [C]//International Symposium on Signal Processing and Information Technology. Bilbao: IEEE, 2017: 410-415.
[本文引用: 2]
[15]
GOODFELLOW I, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial networks [C]// Advances in Neural Information Processing Systems . Montreal: MIT Press, 2014: 2672-2680.
[本文引用: 1]
[16]
ZHU J, PARK T, ISOLA P, et al. Unpaired image-to-image translation using cycle-consistent adversarial networks [C]// International Conference On Computer Vision. Venice: IEEE, 2017: 2242-2251.
[本文引用: 1]
[17]
HUANG G, LIU Z, LAURENS V, et al. Densely connected convolutional networks [C]// Conference on Computer Vision and Pattern Recognition. Honolulu: IEEE, 2017: 2261-2269.
[本文引用: 1]
[18]
CHEN Y, LI J, XIAO H, et al. Dual path networks [C]// Conference on Neural Information Processing Systems. Long Beach: IEEE, 2017: 1-9.
[本文引用: 1]
[19]
JIE H, LI S, GANG S, et al Squeeze-and-excitation networks
[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence , 2017 , 42 (99 ): 2011 - 2023
[本文引用: 1]
[21]
VAHADANE A, PENG T, ALBARQOUNI S, et al. Structure-preserved color normalization for histological images [C]// International Symposium on Biomedical Imaging. New York: IEEE, 2015: 1012-1015.
[本文引用: 1]
Cancer Statistics in China, 2015
1
2016
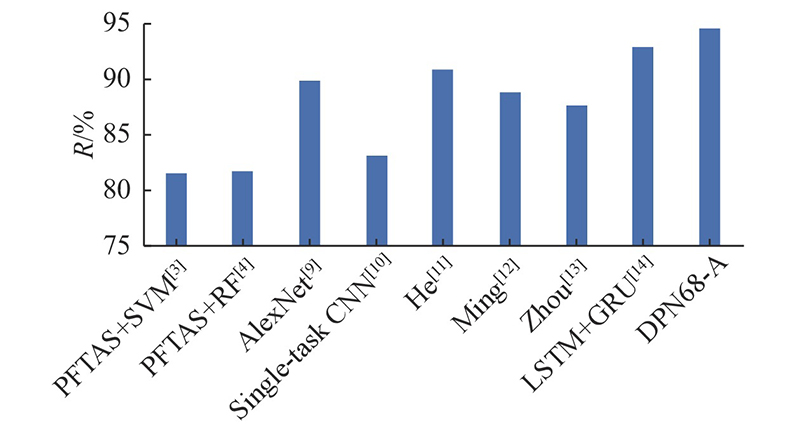
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
Computer-aided diagnosis of breast cancer based on fine needle biopsy microscopic images
1
2013
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
Automatic classification of tissue malignancy for breast carcinoma diagnosis
2
2018
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
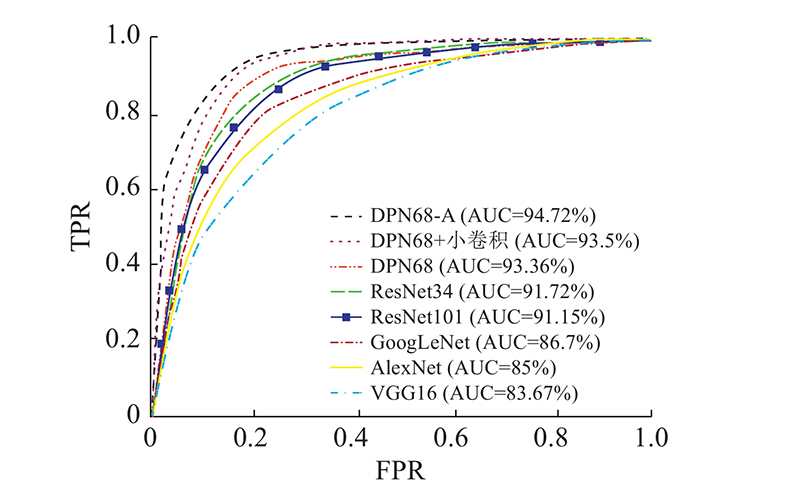
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
Histopathological breast-image classification using concatenated R-G-B histogram information
2
2019
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
Automatic cell nuclei segmentation and classification of breast cancer histopathology images
1
2016
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
A dataset for breast cancer histopathological image classification
1
2016
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
Breast cancer histopathology image analysis: a rcview
1
2014
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
Deep learning in medical image analysis
1
2017
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
2
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
2
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于深度学习的乳腺癌病理图像自动分类
2
2018
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于深度学习的乳腺癌病理图像自动分类
2
2018
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于多尺度通道重校准的乳腺癌病理图像分类
2
2020
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于多尺度通道重校准的乳腺癌病理图像分类
2
2020
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于卷积神经网络的乳腺癌组织病理图像分类
2
2020
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
基于卷积神经网络的乳腺癌组织病理图像分类
2
2020
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
2
... 乳腺组织病理检查是诊断乳腺疾病的最权威标准[1 ] . 传统辅助诊断通过边缘检测的方法分割细胞核[2 ] ,以辅助病理医生诊断. 基于机器学习的方法,如支持向量机[3 ] 、随机森林[4 ] ,一般采用人工提取的特征进行建模和分类[5 -6 ] . 病理图像往往存在较大的差异性[7 ] ,特征提取依赖于很强的专业知识,难以提取完备,因而分类精度较低. 深度学习方法能够克服人工提取特征的局限性,自动抽取复杂的非线性特征,在病理图像的分类中逐步得到广泛应用[8 ] . 在BreakHis数据集上,Spanhol等[9 ] 基于AlexNet模型,结合最大融合方法进行分类,患者级别和图像级别的分类准确度分别为90%和85.6%. Bayramoglu等[10 ] 训练了2个CNN(convolutional neural network)网络,单任务CNN模型用于预测乳腺癌肿瘤的良恶性,多任务CNN用来预测恶性子类,在患者级别上的二分类和四分类准确率分别为83.25%和82.13%. 何雪英等[11 ] 基于GoogLeNet,引入微调学习机制,在患者级别上二分类准确率平均为91%. 明涛等[12 ] 提出多尺度通道重校准模型msSE-ResNet(multi-scale channel squeeze-and-excitation),良恶性分类精度为88.87%. 邹文凯等[13 ] 提出BN-Inception(batch normalization-inception)模型,在训练时不区分放大倍数,在40×病理图片上的分类准确率为87.79%. Nahid等[14 ] 使用频域信息提取特征,利用长短期记忆神经网络(long short term memory,LSTM)及门控循环单元(gated recurrent unit, GRU)进行分类,分类准确率为93.01%. 这些研究证明了基于深度学习方法进行病理图像分类的有效性. ...
... 为了进一步证明所提出的DPN68-A的性能,与PFTAS + SVM[3 ] 和PFTAS+RF方法[4 ] 、AlexNet方法[9 ] 、Single-task CNN方法[10 ] 、改进的深度卷积神经网络模型[11 ] (这里命名为He)、多尺度重校准模型[12 ] (这里命名为Ming)、BN-Inception分类模型[13 ] (这里命名为Zhou)以及LSTM+GRU分类模型[14 ] 进行对比,在患者级别上的准确率结果如图10 所示. ...
1
... 生成对抗网络(generative adversarial network,GAN)[15 ] 目前在图像生成方面已得到广泛应用. 基本结构含有生成器和判别器,通过生成器和判别器的博弈,不断优化损失函数,生成与真实数据十分相似的伪数据. Zhu等[16 ] 提出的Cycle-GAN是在GAN的基础上发展的环形网络结构,可以实现无配对图像之间的风格迁移,在保证生成图像颜色改变的同时保证与原始图像的细节特征保持一致. ...
1
... 生成对抗网络(generative adversarial network,GAN)[15 ] 目前在图像生成方面已得到广泛应用. 基本结构含有生成器和判别器,通过生成器和判别器的博弈,不断优化损失函数,生成与真实数据十分相似的伪数据. Zhu等[16 ] 提出的Cycle-GAN是在GAN的基础上发展的环形网络结构,可以实现无配对图像之间的风格迁移,在保证生成图像颜色改变的同时保证与原始图像的细节特征保持一致. ...
1
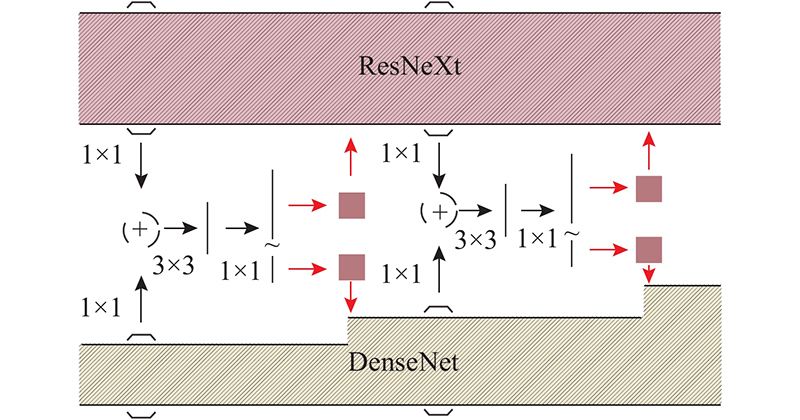
... DPN是基于ResNeXt和DenseNet[17 ] 提出的双路径结构网络. 它融合了ResNeXt和DenseNet的优点,将每一层的输出从相加改为相并联,实现了每一层都能直接得到之前所有层的输出,使得模型对特征的利用更加充分. ...
1
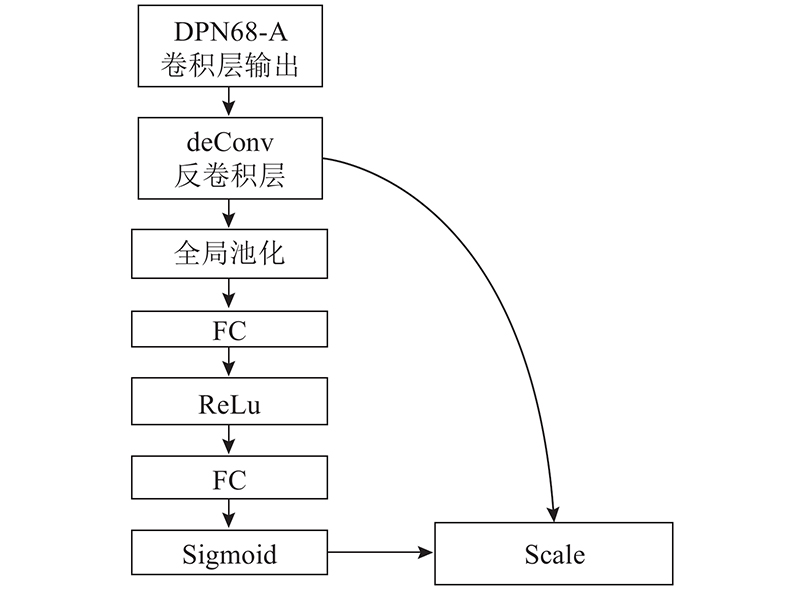
... 注意力机制源于对人类视觉的研究. 人类在生活中为了合理分配有限的视觉信息处理资源,需要选择特定部分来重点关注. 在神经网络中,可以通过添加注意力层,给输入图像的不同部分分配不同的权重[18 ] ,提高良恶性分类精度. 注意力层的结构如图4 所示. ...
Squeeze-and-excitation networks
1
2017
... [19 ] . ...
A survey on image data augmentation for deep learning
1
2019
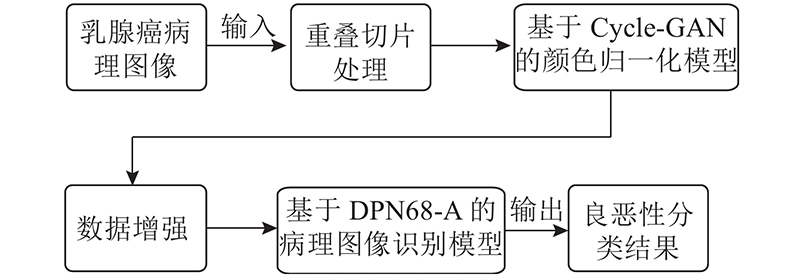
... 3)针对不均衡数据问题对其进行数据增强. 通过翻转、旋转、微调亮度和对比度等方法进行数据扩充[20 ] ,使得良性切片个数与恶性切片个数达到基本平衡. ...
1
... 颜色归一化对比实验在病理图像40×数据集中,随机选取300张良性图像与300张恶性图像作为实验数据. 按照7∶3构造训练集和测试集,训练集和测试集中患者样本无交叉. 通过实验,对比不进行颜色归一化处理、采用Vahadane方法进行归一化处理[21 ] 以及采用Cycle-GAN模型进行归一化处理对检测精度的影响. 检测模型采用本文所提的DPN68-A,基于ImageNet-5K预训练后进行参数微调. 使用预训练参数迁移学习迭代100次,计算最终的准确率作为评价指标. 实验结果如表3 所示. ...