大银鱼(Protosalanx chinensis)是我国大水面渔业的传统经济品种,生活史短,仅为一年生,采用R型繁殖策略,因此,大银鱼早期资源对其种群规模起决定性作用[1-2]。近年来,中国大银鱼资源总体呈下降趋势[3],实施人工放流是有效恢复大银鱼种群的技术手段[2]。大银鱼人工规模化苗种培育技术与其他品种拥有的成熟技术相比,明显存在着技术进程上的缺失。在大银鱼苗种培育过程中,饲料无法使其开口,必须在大银鱼发育阶段的不同时期适当引入不同的天然饵料以保证仔鱼培育需求[4],因此,其肠道微生物在大银鱼发育过程中的消化辅助作用不容忽视。大银鱼的肠道是各种微生物共存的消化器官,肠道微生物、肠道黏膜和免疫系统共同促进大银鱼的消化过程。大银鱼的消化系统十分简单,消化道仅由食道和肠组成,无胃结构,肠道在体内呈直管状,无弯曲,也没有明显的前、中、后之分[1]。前期研究观察发现,大银鱼孵化后1~3 d为内源性营养期,4~9 d为混合性营养期,发育10 d后,大银鱼便可摄食与成体食谱相近的大型桡足类物种。大银鱼的快速发育过程除自身消化器官参与外,还可能与微生物的辅助消化有关。目前,对大银鱼早期发育的研究还不足,对其微生物群落的研究尚未见报道。
在鱼类的早期发育阶段,宿主和微生物群落之间就已经发生了复杂的相互作用[5]。在胚胎阶段,微生物在受精卵表面定植[6]。刚孵化出来的幼鱼开始进食后,其肠道系统暴露在环境中并迅速被细菌定植[7]。肠道菌群的定植一方面有利于刺激免疫系统[8],提高幼鱼对病毒感染的抵抗力;另一方面可以促进鱼类的营养代谢,特别是胆固醇代谢和运输过程[9]。大银鱼开口摄食后,胆汁酸生物合成和代谢、胆固醇分解代谢过程等通路显著富集[10],原因可能是受到了肠道菌群的刺激。另外,鱼类胚胎阶段和幼鱼阶段的一些微生物被认为是导致鱼苗死亡的因素之一[11-12]。针对大银鱼孵化率低和早期生存能力低的情况,研究大银鱼早期发育阶段微生物群落的动态变化,有助于分析大银鱼摄食前后的优势菌群,促进大银鱼开口阶段新型益生菌的开发,从而推动大银鱼人工苗种培育技术的进步。基于此,本研究采用16S rRNA高通量测序技术分析大银鱼早期发育阶段的微生物组成,通过功能预测探讨微生物区系的潜在功能变化,并通过微生物关联网络筛选出大银鱼摄食前后的关键细菌;最后,通过构建大银鱼摄食前后的微生物群落和基因关联网络,挖掘与免疫和代谢基因重点相关的菌属。
1 材料与方法
1.1 样品采集
本研究选择了大银鱼早期发育阶段的5个时期(表1):胚胎期(心跳期,XT)、内源性营养期(采样时间为孵化后第1天,记为H1)、混合性营养期(孵化后第4天,H4)、开口摄食期(孵化后第7天,H7)、外源性营养期(孵化后第10天,H10)。使用Leica DM IL LED倒置荧光显微镜观察,并使用Leica DFC450 C成像系统收集每个时期的图像,结果如图1所示。然后无菌采集大银鱼卵和幼鱼,放入冻存管中,并立即放入液氮中临时保存,采集完成后转移至-80 ℃冰箱中保存,每组重复3次,共采集15个样本。本研究中采用的微生物样品与前期试验的转录组样品为同批次样品[10]。转录组数据已上传至NCBI数据库(
表1 样本详情
Table 1
| 时期 Period | 采样时间 Sampling time | 肠道内食物 Intestinal food | 卵黄囊 Yolk sac |
|---|---|---|---|
| XT | 心跳期 | 无 | |
| H1 | 孵化后第1天 | 无 | 约占鱼体积的3/4 |
| H4 | 孵化后第4天 | 零星出现 | 约占鱼体积的1/2 |
| H7 | 孵化后第7天 | 轮虫 | 约占鱼体积的1/4 |
| H10 | 孵化后第10天 | 轮虫及桡足类 | 卵黄囊消失 |
XT:胚胎期;H1:内源性营养期;H4:混合性营养期;H7:开口摄食期;H10:外源性营养期。下同。
XT: Embryonic period; H1: Endogenous nutrition period; H4: Mixed nutrition period; H7: Open feeding period; H10: Exo-genous nutrition period. The same as below.
图1
图1
大银鱼早期发育阶段5个时期的示意图
Fig. 1
Schematic diagrams of five periods at the early developmental stage of P. chinensis
1.2 DNA提取和扩增
所有样本DNA均用动物组织DNA快速提取试剂盒(美国Omega Bio-Tek公司)进行提取。采用Qubit 4荧光定量仪检测DNA浓度。采用1.5%琼脂糖凝胶电泳检测DNA完整性,电压设置为150 V,时间设置为25 min。然后选择合格的样品进行聚合酶链反应(polymerase chain reaction, PCR)扩增。取30 ng合格的基因组DNA样本,使用引物341F(5
1.3 数据处理与分析
使用QIIME 2进行微生物组生物信息学分析[13-14]。使用DADA2生成扩增子序列变异(amplicon sequence variant, ASV)[15],然后通过MAFFT将所有ASVs对齐[16],再使用FastTree2构建系统发育树[17]。使用q2-feature-classifier对参考序列的ASVs进行分类[18]。将所有样本稀释为每个样本9 213个序列后,使用q2-diversity评估微生物组的α多样性和非度量多维尺度(non-metric multidimensional scaling, NMDS)的β多样性。使用Vegan v2.5-7进行排列多变量方差分析。使用PICRUSt2进行微生物功能预测。使用R v4.1.2对全部数据进行可视化,使用Pheatmap v1.0.12绘制热图,使用ggClusterNet构建网络和Hub ASVs[19],其他图片均由ggplot2 v3.3.6绘制。
2 结果与分析
2.1 微生物多样性
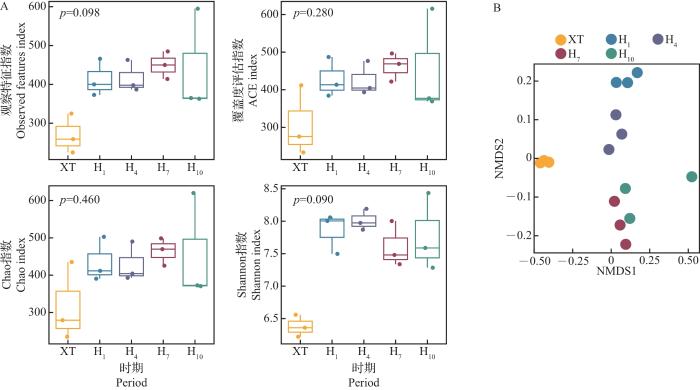
图2
图2
5个时期微生物群落的 α 多样性和 β 多样性分析
A. α多样性分析的箱线图(使用Kruskal-Wallis方法检验各时期间差异的显著性);B.基于Bray-Curtis距离的非度量多维尺度的β多样性分析。
Fig. 2
α and β diversity analyses of microbial communities of five periods
A. Box plots of α diversity analysis (the Kruskal-Wallis method was used to test the significant differences between periods); B. β diversity analysis of NMDS based on Bray-Curtis distance.
2.2 微生物组成
从门水平(图3)上看,大银鱼早期发育过程中的微生物群落主要由变形菌门(Proteobacteria)、拟杆菌门(Bacteroidota)和厚壁菌门(Firmicutes)3个菌群组成。变形菌门在H1和H4中的相对丰度均超过50%,均为63.00%,在其他时期中的相对丰度也超过25%(XT为29.13%,H7为47.10%,H10为38.12%)。拟杆菌门在XT中的相对丰度最高,达到63.00%,显著高于其他时期(H1为11.00%,H4为21.00%,H7为37.21%,H10为31.07%)。厚壁菌门在XT中相对丰度较低,为0.51%,但在其他时期相对丰度增加(H1为18.00%,H4为5.50%,H7为8.61%,H10为16.10%)。综上可知,大银鱼胚胎期以拟杆菌门为主,内源性营养期以变形菌门为主,混合性营养期到外源性营养期均以变形菌门和拟杆菌门为主。
图3
图3
5个时期的微生物组成(门水平)
1~3代表相同时期下的3个重复样本。图4同。
Fig. 3
Microbial compositions of five periods (phylum level)
1-3 represent three replicate samples under the same period. The same as Fig. 4.
从属水平(图4)上看,黄杆菌属(Flavobacterium)在XT中相对丰度较高,达到42.13%,其次是金黄杆菌属(Chryseobacterium)。金黄杆菌属在其他时期的相对丰度均低于1.50%(H1为1.20%,H4为0.56%,H7为0.55%,H10为0.71%)。假单胞菌属(Pseudomonas)在各时期均稳定存在,在H1和H4中相对丰度较高(XT为8.50%,H1为17.01%,H4为15.00%,H7为9.50%,H10为9.22%)。大银鱼开始摄食后,弯曲杆菌属(Flectobacillus)的相对丰度增加(H7为23.03%,H10为19.11%),而摄食前其相对丰度较低(XT为0.40%,H1为1.10%,H4为0.38%)。此外,短波单胞菌属(Brevundimonas)和不动杆菌属(Acinetobacter)在大银鱼摄食前后的整体微生物群落中占比均为2.00%以上。综上所述,大银鱼胚胎期的主要菌群是黄杆菌属和金黄杆菌属,内源性营养期的主要菌群是假单胞菌属,混合性营养期的主要菌群是黄杆菌属和假单胞菌属,开口摄食期和外源性营养期的主要菌群是弯曲杆菌属和假单胞菌属。
图4
图4
5个时期的微生物组成(属水平)
Fig. 4
Microbial compositions of five periods (genus level)
2.3 微生物功能
本研究使用PICRUSt2预测微生物群落的功能,并用Kruskal-Wallis方法检验差异的显著性。共筛选出74条京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路,其中57条属于代谢通路(图5),17条属于环境信息处理、遗传信息处理、细胞过程等通路。如图5所示,在大银鱼摄食前后,脂质代谢中的亚油酸代谢、类固醇生物合成通路,以及辅助因子和维生素代谢中的泛酸和辅酶A(CoA)生物合成、生物素代谢通路显著富集。能量代谢中的4条通路,以及聚糖生物合成和代谢中除肽聚糖生物合成外的其余4条通路随着大银鱼的发育进程而逐渐减少富集,而在H1和H4中,异生素的生物降解和代谢中的多条通路显著富集。
图5
图5
5个时期微生物群落的KEGG功能分析
每个热图代表1个KEGG通路二级分类。每个色块代表每个时期的功能丰度平均值并以行为中心进行缩放,红色代表丰度高,蓝色代表丰度低。
Fig. 5
KEGG functional analysis of microbial communities of five periods
Each heat map represents a KEGG pathway secondary classification. Each color block represents the mean value of functional abundance of each period and is centered and scaled by row. Red represents a high abundance, and blue represents a low abundance.
2.4 微生物关联网络
图6
图6
5个时期的微生物关联网络
A.微生物模块化网络(每个节点代表1个ASV,节点大小代表ASV的平均相对丰度。红色线段表示节点间呈负相关,蓝色线段表示节点间呈正相关);B.模块化网络的Hub节点分析(Hub得分越高,与其他节点的关联程度就越高)。
Fig. 6
Microbial association networks of five periods
A. Microbial modular networks (each node represents an ASV, and the node size represents the average relative abundance of ASV. The red line segment indicates a negative correlation between the nodes, and the blue line segment indicates a positive correlation between the nodes); B. Analysis for Hub nodes of the modular network (the higher the Hub score, the higher the degree of association with other nodes).
2.5 微生物群落与免疫和代谢基因的关联网络
图7
图7
5个时期微生物群落基因的KEGG注释及分类结果
Fig. 7
KEGG annotation and classification results of genes in microbial communities of five periods
为分析微生物群落与免疫相关基因的关联性,利用免疫系统通路中的2 088个基因与相对丰度排名前10%的ASVs构建关联网络。节点关联度(igraph degree)大于10的ASVs节点有9个,其中6个ASVs节点来自弯曲杆菌属,其余3个ASVs节点分别来自Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium、金黄杆菌属和黄杆菌属(图8)。关联网络显示,弯曲杆菌属的ASVs与35个基因呈负相关,与5个基因呈正相关;黄杆菌属的ASV与9个基因呈负相关,与34个基因呈正相关;Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium的ASV与所有关联的18个基因均呈负相关;而金黄杆菌属的ASV与关联的13个基因均呈正相关,仅与1个基因呈负相关。
图8
图8
微生物群落与免疫通路相关基因的关联网络
每个节点代表1个ASV或1个基因,节点的大小代表节点关联度。节点标签表示ASV所属的细菌属名。红色线段表示节点间呈负相关,蓝色线段表示节点间呈正相关。
Fig. 8
Association networks of microbial community and immune pathway related genes
Each node represents an ASV or a gene, and the node’s size represents the igraph degree. Node label indicates the genus name to which the ASV belongs. The red line segment indicates a negative correlation between the nodes, and the blue line segment indicates a positive correlation between the nodes.
为分析微生物群落与代谢相关基因的关联,利用代谢通路中的1 528个基因与相对丰度排名前10%的ASVs构建关联网络,结果如图9所示。节点关联度大于5的ASVs节点共有13个,其中弯曲杆菌属有7个,黄杆菌属有2个,Allorhizobium-Neorhizo-bium-Pararhizobium-Rhizobium有1个,金黄杆菌属有1个(图9B)。弯曲杆菌属的ASVs与20个基因呈负相关,与4个基因呈正相关。黄杆菌属的ASVs与15个基因呈正相关,与11个基因呈负相关。在节点关联度大于5的基因节点中,只有ENO与弯曲杆菌属的ASVs呈正相关,其余基因节点与弯曲杆菌属的ASVs呈负相关(图9C~D)。维恩图(图9A)显示,有46个基因同时参与了脂质代谢、碳水化合物代谢、氨基酸代谢相关通路。根据这46个基因和相对丰度排名前10%的ASVs构建的关联网络,弯曲杆菌属中有4个ASVs与HADHA基因呈负相关,黄杆菌属中有1个ASV与ALDH7A1基因呈负相关。
图9
图9
微生物群落与代谢通路相关基因的关联网络
A.碳水化合物代谢、氨基酸代谢、脂质代谢相关基因的维恩图;B.微生物群落和代谢通路相关基因关联网络(每个节点代表1个ASV或基因,节点的大小代表节点关联度。节点标签表示细菌属名或基因名称。红色线段表示节点间呈负相关,蓝色线段表示节点间呈正相关);C.节点关联度大于5的ASVs节点的热图;D.节点关联度大于5的基因节点的热图。
Fig. 9
Association networks of microbial community and metabolic pathway related genes
A. Venn diagram about genes belonging to carbohydrate metabolism, amino acid metabolism, and lipid metabolism; B. Association network of microbial community and metabolic pathway related genes (each node represents an ASV or a gene, and the node’s size represents the igraph degree. Node label indicates genus name or gene name. The red line segment indicates a negative correlation between the nodes, and the blue line segment indicates a positive correlation between the nodes); C. Heat map of ASV nodes with igraph degree greater than 5; D. Heat map for gene nodes with igraph degree greater than 5. ANPR: Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium.
3 讨论与结论
3.1 大银鱼早期发育阶段不同时期的微生物群落特征
研究鱼类微生物对改善鱼类生存环境条件具有重要意义,尤其在鱼类开始摄食阶段,幼鱼的组织器官尚未完全发育,容易受到外部环境的影响,导致幼鱼的高死亡率。有研究表明,在鱼类早期发育阶段使用适量的微生物(益生菌)会使得宿主免受病原体感染,生存率提高,这类微生物已应用于鱼虾的规模化培育[20]。本研究通过16S rRNA基因测序技术获得了大银鱼早期发育阶段各时期较完整的微生物信息。
幼鱼微生物群落的组成随着年龄的增长而发生变化,其多样性也会随年龄的增长而增加,表明微生物组在幼鱼的发育进程中实现了演替[21]。大银鱼早期发育阶段的微生物多样性呈上升趋势,且胚胎期(XT)和幼鱼阶段(H1、H4、H7、H10)菌群分群明显,可能与开始摄食或者外部环境中的细菌影响等因素有关。大银鱼在内源性营养期主要以变形菌门为主,而在混合性营养期和外源性营养期均以变形菌门和拟杆菌门为主,且从混合性营养期到外源性营养期过程中变形菌门占比逐渐减少,拟杆菌门逐渐增多。大银鱼早期发育阶段的微生物组成与生态习性相近的鳕鱼幼鱼的微生物组成相似度很高,而鳕鱼幼鱼的微生物群落与其生存的水环境和活饲料中的微生物群落之间差异较大[22]。并且,鱼类幼鱼中的细菌变化过程很复杂,一般认为取决于卵、饲养水和活饲料中的微生物[23]。在内源性营养期,大银鱼幼鱼发育所需的营养物质完全来自卵黄囊,此时的微生物群落可能是大银鱼胚胎阶段特殊的共生微生物群落。在混合性营养期大银鱼开始摄食,在外源性营养期大银鱼完全依赖外界环境中的食物摄取营养,且幼鱼摄食种类逐渐发生变化。开始摄食后,大银鱼的变形菌门逐渐变少,拟杆菌门逐渐变多,说明摄食可能会导致大银鱼微生物群落发生演替。关于大银鱼摄食前后的菌群来源需要结合水环境中的微生物群落作进一步分析。
在属水平上,大银鱼胚胎期的黄杆菌属占比较高,后期黄杆菌属占比较低。研究表明,黄杆菌属中的嗜铬黄杆菌是一种与鱼苗相关的病原体,可引起虹鳟鱼苗综合征和细菌性冷水病[24-25],细菌性冷水病通常发生在10 ℃左右[25],而大银鱼胚胎发育的最佳温度为2~8 ℃[1]。另一种在大银鱼胚胎期高度富集的菌群是金黄杆菌属,它与黄杆菌属同属黄杆菌科。金黄杆菌属和黄杆菌属被认为存活于鲑鱼发育早期的卵和鱼苗中,很难被杀灭[26],但黄杆菌属在鱼类胚后发育阶段是否表现为致病性,仍需进一步研究。本研究筛选出的大银鱼早期发育阶段各时期的关键微生物是假单胞菌属。假单胞菌属在环境中广泛分布,大多数研究认为,假单胞菌属是鱼类重要的条件致病菌[27]。另外一些研究则表明,假单胞菌属可能是一种益生菌,例如荧光假单胞菌有助于抑制虹鳟鱼的水霉病[28],在饲料中添加铜绿假单胞菌VSG2可增强鱼类的免疫反应和抗氧化能力,并提高鱼类对嗜水气单胞菌感染的耐受性[29]。在与大银鱼生态习性相似的鳕鱼幼鱼和大马哈鱼幼鱼中,假单胞菌属均为优势菌群[21,30],假单胞菌属可能对冷水性鱼的幼鱼有着不可代替的作用。
进一步研究假单胞菌属、黄杆菌属和金黄杆菌属与大银鱼的相互作用将有助于提高对大银鱼早期发育的认识。
3.2 微生物与大银鱼免疫基因和代谢基因的关联分析
鱼类的微生物群落参与重要的生物学功能,如营养代谢、免疫和其他生理过程[8-9]。本研究基于关联网络方法筛选了与免疫和代谢相关的菌属。与大银鱼免疫基因相关的微生物主要是弯曲杆菌属,其次是Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium、黄杆菌属和金黄杆菌属。微生物可以通过调节胃肠道的免疫系统间接提高幼鱼的免疫力[31]。目前对弯曲杆菌属在鱼类中的功能了解有限,尤其在免疫方面,仅初步了解到弯曲杆菌属及其分泌的鞘脂在调节虹鳟鱼黏膜免疫系统中发挥着关键作用[32]。Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium是固氮细菌,主要与植物生长有关,也有研究发现它的早期定植会使鸡的肠道中释放更多的血清脂多糖,诱导炎症细胞因子的表达,引起肠道炎症[33]。在淡水鱼类中,黄杆菌属多被认为是病原体,会破坏鱼类免疫系统,以及造成鳃、皮肤、鳍和尾巴的严重组织损伤[34]。马西黄杆菌(Chryseobacterium massiliae)可以保护斑马鱼幼鱼免受柱状黄杆菌(Flavobacterium columnare)的侵害[34],说明微生物之间也可以相互制约。在大银鱼早期发育过程中,尤其在开始摄食后,弯曲杆菌属的占比增加,黄杆菌属的占比明显下降,既可能是受到摄食的影响,也可能是微生物之间的相互作用导致的,从而引起系统的免疫反应,使致病菌减少,以及鱼苗的存活率提高。
与大银鱼代谢基因相关的微生物同样主要是弯曲杆菌属,其次是Allorhizobium-Neorhizobium-Pararhizobium-Rhizobium、黄杆菌属和金黄杆菌属。微生物在营养代谢中起着至关重要的作用,它们参与营养代谢,特别是胆固醇的代谢和运输,而前期转录组研究结果也显示,大银鱼开始摄食后,胆汁酸生物合成和代谢、胆固醇分解代谢过程等通路显著富集[10]。上述3种菌属在鱼类中的代谢作用尚不清楚,但其他杆菌,例如拟杆菌属、梭状芽孢杆菌属、丙酸杆菌属、梭杆菌属、乳杆菌属能降解蛋白质,促进蛋白质的代谢,合成多种维生素,促进自身新陈代谢以及维持宿主生理机能,同时具有水解酶编码基因,可以促进胆汁酸的合成,有助于脂质消化[35]。结合以上研究可知,弯曲杆菌属可能也具备这些功能,即促进大银鱼胆汁酸代谢和胆固醇分解代谢。与免疫和代谢基因表达最为相关的均为弯曲杆菌属等节点菌属。虽然没有研究表明弯曲杆菌属等与本研究筛选的基因表达直接相关,但弯曲杆菌属与大银鱼免疫和代谢的关系同样值得进一步研究。
本研究首次获得了大银鱼早期发育过程中的微生物演替信息,并筛选出关键菌属,以及与免疫和代谢重点相关的节点菌属。由于大银鱼规模化培育技术尚未完善,缺少实践应用,并且大银鱼于冬季繁殖,环境条件匮乏,在其早期发育阶段不同时期投喂适宜的益生菌有助于提高大银鱼的早期存活率。本研究筛选的微生物可为开发适用于培育大银鱼的益生菌提供基础信息,为开发大银鱼苗种规模化培育技术手段提供理论依据。
参考文献
大银鱼生物学与渔业生态学研究进展
[J].
Biology and fishery ecology of Protosalanx chinensis: a review
[J].DOI:10.11964/jfc.20200312210 [本文引用: 3]
水库大银鱼种群变动规律及增殖利用初步研究
[J].
Preliminary study on variation pattern of Protosalanx chinensis population and its proliferation and utilization
[J].DOI:10.3969/j.issn.1004-2091.2004.03.018 [本文引用: 2]
Threats and protection policies of the aquatic biodiversity in the Yangtze River
[J].
大银鱼仔鱼的人工培育技术探究
[J].
Research on artificial breeding technology of Protosalanx chinensis larvae
[J].DOI:10.19415/j.cnki.1673-890x.2021.29.080 [本文引用: 1]
Gnotobiotic models: powerful tools for deeply understanding intestinal microbiota-host interactions in aquaculture
[J].
Effects of host genetics and environment on egg-associated micro-biotas in brown trout (Salmo trutta)
[J].
The intestine of artificially bred larval turbot (Scophthalmus maximus) contains a stable core group of microbiota
[J].
Effect of germ-free rearing environment on gut development of larval sea bass (Dicentrarchus labrax L.)
[J].
Gnotobiotic zebrafish reveal evolutionarily conserved responses to the gut microbiota
[J].
Transcriptome sequencing analysis reveals dynamic changes in major bio-logical functions during the early development of clearhead icefish, Protosalanx chinensis
[J].
Use of gnotobiotic zebrafish to study Vibrio anguillarum pathogenicity
[J].
Microbial colonization induces dynamic temporal and spatial patterns of NF-κB activation in the zebrafish digestive tract
[J].
Repro-ducible, interactive, scalable and extensible microbiome data science using QIIME 2
[J].
泌乳中期荷斯坦奶牛瘤胃细菌群落组成与多样性变化
[J].
Changes in the com-position and diversity of the rumen bacterial community in mid-lactation Holstein cows
[J].DOI:10.3785/j.issn.1008-9209.2022.06.291 [本文引用: 1]
DADA2: high-resolution sample inference from Illumina amplicon data
[J].
MAFFT: a novel method for rapid multiple sequence alignment based on fast Fourier transform
[J].
FastTree2—approxi-mately maximum-likelihood trees for large alignments
[J].
Optimizing taxonomic classification of marker-gene amplicon sequences with QIIME 2’s q2-feature-classifier plugin
[J].
ggClusterNet: an R package for microbiome network analysis and modularity-based multiple network layouts
[J].
Probiotics in animal nutrition and health
[J].
Whole gut microbiome composition of damselfish and cardinalfish before and after reef settlement
[J].
Selection in the host structures the microbiota associated with developing cod larvae (Gadus morhua)
[J].
Progress in fish gastrointestinal microbiota research
[J].
A review of Flavobacterium psychrophilum biology, clinical signs, and bacterial cold water disease prevention and treat
[J].
Bacterial coldwater disease of fishes caused by Flavobacterium psychrophilum
[J].
Flavobacteria colonizing the early life stages of hatchery-incubated Chinook salmon Oncor-hynchus tshawytscha (Walbaum 1792) are markedly diverse
[J].
First occurrence and whole-genome comparison of Pseudomonas haemolytica isolated in farmed rainbow trout
[J].
Possible mechanisms of action of two Pseudomonas fluorescens isolates as probiotics on saprolegniosis control in rainbow trout (Oncorhynchus mykiss Walbaum)
[J].
Effects of dietary heat-killed Pseudomonas aeruginosa strain VSG2 on immune functions, antioxidant efficacy, and disease resistance in Cyprinus carpio
[J].
16S rDNA-based analysis of dominant bacterial populations associated with early life stages of coho salmon (Oncorhynchus kisutch)
[J].
Role of gastrointestinal microbiota in fish
[J].
Symbiont-derived sphingolipids modulate mucosal homeostasis and B cells in teleost fish
[J].
Chicken jejunal microbiota improves growth performance by mitigating intestinal inflammation
[J].
Mining zebrafish microbiota reveals key community-level resistance against fish pathogen infection
[J].
A review of metabolic potential of human gut microbiome in human nutrition
[J].