睡眠帮助人们消除疲劳、恢复体力和精力, 睡眠质量的好坏影响人的身心健康和工作生活.人每天都需要进行睡眠, 但真正了解自己睡眠状况的人并不多.一个重要的原因在于目前还没有既简便又经济的方法让人们了解自己的睡眠质量.
睡眠分期是进行睡眠质量评价的重要依据, 但已有的睡眠分期方法大都以脑电信号为主.当前最广泛使用的是利用多导睡眠图仪(PSG)采集脑电、心电、肌电等信号, 最后由人工完成分期.该设备一方面本身比较复杂且需要专业人员在特定的睡眠测试场所进行, 另一方面需要在受试者身上粘贴许多的传感器, 给测试者带来较大的生理和心理负荷, 需要受试者有一定的耐受性, 对自然睡眠造成了严重侵扰.并且由于价格昂贵, 操作复杂, 该设备不适应家庭使用, 无法向公众推广.
本文提出基于脉率变异性的睡眠分期方法.采用血氧指套采集脉搏信号, 再经过脉搏信号提取单个脉搏间期并进行特征提取.对于提取的特征进行频域分析, 根据分析结果进行睡眠分期的方法, 可以在居家环境和接近无侵扰睡眠的情况下实现睡眠的自动分期, 并且达到一定的准确程度.
1 相关工作目前的睡眠分期方法一般可归为3类.
第一类是基于多导睡眠监测仪(PSG).利用在人体上粘贴多个电极的方法获取受试者的脑电图(EEG)、眼动图(EOG)、还有某些骨骼肌如下颌肌的肌电图(EMG), 由人工判读标示完成睡眠分期.具体过程是从睡眠开始以30 s为一段进行分屏, 利用给出的各睡眠期中α和β波所占的比例、肌电的活动、眼球的运动等特征, 连续进行睡眠分期[1].这种方法具有较高的精度, 为当前睡眠分期的主流方法, 但这种方法所需的仪器比较昂贵, 只有专业的人员才能使用, 还需要在专门的睡眠实验室进行, 受试者身上需要粘贴多个电极, 在一定程度上对自然睡眠造成破坏.
第二类方法是使用多种生理参数实现睡眠分期.杨军等[2, 3]在无电极、无约束的自然状态下获取心动周期、呼吸波周期、体动指数, 运用人工智能, 从多个生理参数中提取睡眠信息, 应用不确定推理的证据理论, 融合计算多项参数中的睡眠信息, 实现对睡眠的自动分期.与PSG的对照试验中醒睡的平均符合率在90%, 基本睡眠结构的平均符合率在75%以上.Migliorini等[4]通过在床上安装8个方向电极的方法取得睡眠数据, 通过分析HRV、呼吸、体动信号, 使用时变自回归模型和离散小波变换模型, 将特征输入到2个分类器, 使用二次和线性的判别式来进行睡眠分期.Hoshiyama等[5-6]采用心率、眼电图等生理参数来进行睡眠过程中快速眼动期(rapid eye movement)的判别.Foussier等[7]基于心率变异性信号中提取的72个特征对睡眠中的觉醒进行判定,提高了睡眠分期的准确程度.第二类方法虽然在用户体验上有了很大的提升, 但睡眠监测的设备昂贵, 精确度与PSG法相比又相对较低.
第三类方法是基于单个生理参数的睡眠分期方法.文献[8-9]仅使用心率信号进行睡眠分期.庄志等[8]利用HMM(hidden Markov model)的模式识别能力, 对每一种睡眠分期状态建立一个HMM, 并用与之对应的RR间期(心电图中相邻心搏间的距离或者说时间)信号序列进行训练, RR间期就是HMM的观察值, 再利用训练后的HMM分析RR间期信号以判断与该RR间期序列对应的睡眠分期类型.Mendez等[9]采用时变自回归模型(time-varying autoregressive model, TVAM)提取心率变异性特征, 使用HMM作为概率分类器, 区分不同的睡眠阶段.Wang等[10]采用基于决策树的支持向量机算法对心电信号进行分析进而对人体睡眠做出分期.笔者使用从胸冲击信号中提取的心率信号, 通过不同睡眠阶段的心率变异性特征实现睡眠分期[11].Long等[12]仅使用心率变异性检测深度睡眠情况.基于单个生理参数进行睡眠分期的方法多使用心率信号, 但心率信号的采集一般仍需要通过贴片的方式, 舒适度并不太理想.
本文从居家环境和经济实用角度出发, 提出一种信号获取容易、操作方法简单、实用性好、适合大众使用的睡眠分期方法.在整个睡眠监测过程中,受试者只需要佩带一只小巧的血氧指套, 对人体的睡眠过程干扰很小(受试者体验), 具有良好的适用性.血氧指套不仅相对廉价, 而且能够准确地获取人体脉搏信号, 在信号的采集过程中对人的自然睡眠影响很小, 使用方法简单易学.通过获取的脉率信号得到脉率间期, 再分析不同睡眠阶段的脉率变异性特征, 实现睡眠分期.
2 实现方法使用血氧指套取得脉搏原始信号, 信号的采样率为100 Hz, 经多分辨率小波变换后, 提取第一层信号并使用滑动窗口获取单个脉搏间期.利用取得的脉搏间期提取脉率变异性特征, 并根据不同睡眠阶段的脉率变异性特征实现睡眠分期.训练过程中使用的睡眠分期是由睡眠专家人工标记的.基于脉率变异性睡眠分期方法的工作流程如图 1所示.
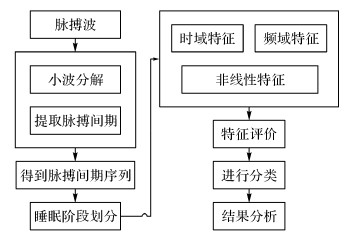
|
图 1 睡眠分期的流程示意图 Fig. 1 Framework of sleep stages classification |
小波分解提取第一层信号[13-14], 从图 2可以看到原始信号中有部分的小毛刺, 如果此时进行脉搏间期提取, 会出现误判.当通过一次小波变换后, 信号就已经非常平滑, 如图 3所示, 此时可以直接用滑动窗口的方法取得脉搏间期值[15-16].
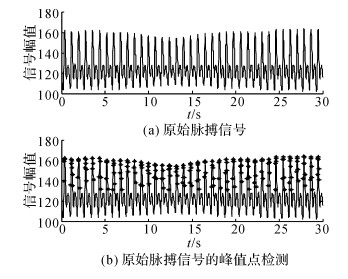
|
图 2 原始信号的峰值点检测 Fig. 2 Peak detection on origin signal |
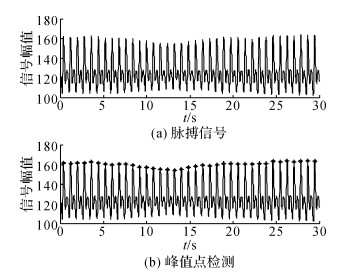
|
图 3 小波变换处理后的峰值点检测 Fig. 3 Peak detection on signal after wavelet transformation |
本实验中使用了一个重迭的滑动窗口, 窗口宽度为90个采样点, 移动步长为70个采样点, 在实际应用中由于数据集的不同, 应根据不同数据特点设置相应的窗口长度和步长.
2.2 特征提取在人体处于静止状态时可以直接使用脉率变异性替代心率变异性, 反映人体的神经变化[12], 而心率变异性分析主要从时域、频域、非线性3个方面进行.Gil等[17]研究发现, 正常人静息状态下的脉率变异性具有与心率变异性相同的特征, 得出在某些情况下可使用脉率变异性替代心率变异性的结论.本文分别从时域、频域、非线性3个方面提取脉率变异性的7个特征.
2.2.1 时域分析时域分析是对连续PP间期(脉率间期)的一个统计, 通过统计指标表达脉率的变化.常用的时域分析统计指标有:均值M:反映PP间期的平均水平;总体标准差S:所有正常PP间期的标准差;差值均方的平方根R:相邻2个PP间期的差值的均方根, 反映HRV快变化部分.
本文采用如下的线性指标:记第i个PP间期为D(PPi), 那么相邻的下一个PP间期为D(PPi+1), N为序列长度.相关指标的计算公式如下:
1) 均值:
| $ M = \frac{1}{N}\sum\limits_{i = 1}^N {D({\rm{P}}{{\rm{P}}_i});} $ |
2) 均值标准差:
| $ S = \sqrt {\frac{1}{N}\sum\limits_{i = 1}^N {{{(D({\rm{P}}{{\rm{P}}_i})-M)}^2}} } ; $ |
3) 均方平方根:
| $ S = \sqrt {\frac{1}{N}\sum\limits_{i = 1}^N {{{(D({\rm{P}}{{\rm{P}}_{i + 1}})-D({\rm{P}}{{\rm{P}}_i}))}^2}} } ; $ |
HRV的频带分为极低频(VLF, 0~0.04 Hz)、低频(LF, 0.04~0.15 Hz)和高频(HF, 0.15~0.40 Hz)[18].其中VLF与热量调节和体液调节相关; LF(L)反映交感和副交感神经对心率的共同调节, 其功率的增加通常被认为是交感神经活动的结果; HF(H)与呼吸有关, 主要反映副交感神经对心率的调控; LF/HF(P)反映交感神经系统与副交感神经系统的状况及其均衡性的变化趋势.对PPi序列进行功率谱分析主要是研究交感和副交感神经在不同睡眠时相的活动情况[18].常用的HRV功率谱分析有2种:一种是快速傅立叶变换(fast Fourier transform, FFT)经典谱估计, 另一种是自回归(autoregressive, AR)模型谱估计[19].AR模型功率谱估计得到的谱曲线平滑, 分辨率高, 容易辨认频谱波峰的中心频率, 尤其在分析短序列数据方面明显优于FFT谱估计, 本文使用AR模型计算脉率变异性(pulse rate variability, PRV)功率谱, 特征选取为L、H、P.
2.2.3 非线性分析脉搏信号具有非线性、非平稳性等特征.使用去趋势波动分析(detrended fluctuation analysis)的方法对健康人不同睡眠时相的脉率进行分析时, 可通过计算不同睡眠阶段脉率的标度指数γ(体现序列的相关性), 研究基于脉率变异性的健康人的睡眠分期[20].由于本文使用10 min长度的数据段来计算不同睡眠阶段的特征均值, 正常的脉率次数大约为500~700次, 通过4 Hz的重采样, 数据段的采样点数大约为2 000~2 800, 在计算γ时使用的片断长度为320~800, 步长为5.
3 数据分析与实验验证实验中共使用了25个健康成年人的整晚睡眠数据(数据由航空医学研究所采得), 其中女性7人, 男性18人, 睡眠分期由专家人工标记完成.在数据的训练过程中使用非重合的数据段计算不同睡眠阶段的特征均值, 长度为10 min.
脉率变异性的特征计算结果如表 1所示, 可得出以下几点结论:
| 表 1 REM睡眠、浅睡眠和深睡眠的PRV频域特征参数对比 Table 1 Parameters comparison among the REM sleep stages, light sleep stages and deep sleep stages |
1) 随着睡眠由快速眼动期(rapid eye movement, REM)到浅睡再到深睡的变化, 不同睡眠阶段中脉搏的均值变化并不是很大, 但方差越来越小, 说明脉率逐渐趋于平稳.
2) 从参数P在REM、浅睡和深睡的不同值可以看出, 副交感神经与交感神经在不断变化, 由于此时期交感神经受到抑制, 副交感神经的作用表现更强, 因此深睡期P值较小.
3) 浅睡、深睡、REM这3个不同睡眠阶段的标度指数γ均大于0.5且小于1.5,表明不同睡眠阶段的脉率变异性中存在着长程相关性.
基于提取的特征, 使用3种分类器对实验数据进行了分类, 从图 4中可以看到, 相对于RotationForest(旋转森林)和ADTree(AD树), NaiveBayes(朴素贝叶斯)具有最佳的分类效果, 准确率达到76%.同时拥有较高的召回率和曲线下面积(area under curve, AUC)值.
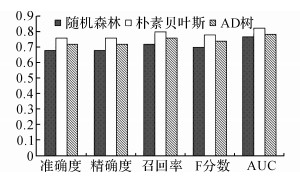
|
图 4 旋转森林、AD树、朴素贝叶斯3种分类器的睡眠分期结果对比 Fig. 4 Results comparison among three different classifiers: RotationForest、ADTree and NaiveBayes |
使用文中所述的方法进行睡眠分期实验, 在仅划分深睡、浅睡和REM三个睡眠阶段而不考虑睡眠过程中的短暂觉醒时, 能够取得最高为76%的准确率, 与文献[8]利用心率变异性进行睡眠分期取得的准确率比较接近.同时, 由于脉搏信号的采集仅要求受试者佩戴一只血氧指套, 相对于获取电信号的贴片方式, 对睡眠的影响非常小, 受试者基本无明显的不适反应.实验结果表明, 该方法不仅能够得到较为准确的睡眠分期, 且对环境和设备的要求较低, 在对睡眠分期结果要求并不十分严格的场合具有良好的适用性, 可满足普通人在家居条件下了解睡眠情况的需要.
| [1] |
王菡侨. 有关美国睡眠医学学会睡眠分期的最新判读标准指南解析[J].
诊断学理论与实践, 2009, 8(6): 575–578.
WANG Han-qiao. Explanation of guidelines for sleep stage classification issued by American of Sleep Medicine[J]. Journal of Diagn Concepts Pract, 2009, 8(6): 575–578. |
| [2] |
杨军, 俞梦孙, 王宏山, 等. 多参数信息融合实现非脑电的睡眠结构分期[J].
中国生物医学工程学报, 2006, 25(3): 315–321.
YANG Jun, YUMeng-Sun, WANG Hong-Shan, et al. A Non_EEG approach to sleep analysis by fusing multi-parameter information[J]. Chinese Journal of Biomedical Engineering, 2006, 25(3): 315–321. |
| [3] |
王海涛, 郑慧君, 曹征涛, 等. 考虑个体特征的非脑电睡眠分期[J].
中国生物医学工程学报, 2010, 29(2): 161–165.
WANG Hai-tao, ZHENG Hui-jun, CAO Zheng-tao, et al. Subject-specific Non-EEG sleep staging algorithm[J]. Chinese Journal of Biomedical Engineering, 2010, 29(2): 161–165. |
| [4] | MIGLIORINI M, BIANCHI A M, NISTICÒ D, et al. Automatic sleep stagging based on ballistocardiographic signals recorded through bed sensors [C] // 2010 Annual International Conference of the IEEE. Buenos Aires: IEEE, 2010: 3273-3276. |
| [5] | HOSHIYAMA M, HOSHIYAMA A. Heart rate variability associated with rapid eye movements during sleep [C] // 2007 Computers in Cardiology. Durham: IEEE, 2007: 689-692. |
| [6] | DENG Z D, POON C S, ARZENO N M, et al. Heart rate variability in pediatric obstructive sleep apnea [C] // Engineering in Medicine and Biology Society, 2006. EMBS'06. 28th Annual International Conference of the IEEE. New York: IEEE, 2006: 3565-3568. |
| [7] | FOUSSIER J, FONSECA P, LONG X, et al. Combining HRV features for automatic arousal detection [C] // Computing in Cardiology 2013. Zaragoza: IEEE, 2013: 1003-1006. |
| [8] |
庄志, 高上凯, 高小榕. 基于心率变异分析的睡眠分期方法[J].
生物医学工程学杂志, 2006, 23(3): 499–504.
ZHUANG Zhi, GAO Shang-kai, GAO Xiao-rong. The sleep staging based on HRV analysis[J]. Journal of Biomedical Engineering, 2006, 23(3): 499–504. |
| [9] | MENDEZ M O, MATTEUCCI M, CERUTTI S, et al. Sleep staging classification based on HRV: time-variant analysis [C] // 2009 Annual International Conference of the IEEE. Minneapolis: IEEE, 2009: 9-12. |
| [10] | WANG J S, SHIH G R, CHIANG W C. Sleep stage classification of sleep apnea patients using decision-tree-based support vector machines based on ECG parameters [C] // Proceedings of 2012 IEEE-EMBS International Conference on Biomedical and Health Informatics. Hong Kong: IEEE, 2012: 285-288. |
| [11] | NI H, ZHAO T, ZHOU X, et al. Analyzing sleep stages in home environment based on ballistocardiography[M]. Switzerland: Springer International Publishing, 2015: 56-68. |
| [12] | LONG X, FONSECA P, HAAKMA R, et al. Automatic detection of overnightdeep sleep based on heart rate variability: a preliminary study [C] // 2014 36th Annual International Conference of the IEEE. Chicago: IEEE, 2014: 50-53. |
| [13] | KEISSAR K, DAVRATH L R, AKSELROD S. Coherence analysis between respiration and heart rate variability using continuous wavelet transform[J]. Philosophical Transactions of the Royal Society A: Mathematical, Physical and Engineering Sciences, 2009, 367(1892): 1393–1406. DOI:10.1098/rsta.2008.0273 |
| [14] | BAR A, PILLAR G, DVIR I, et al. Evaluation of a portable device based on peripheral arterial tone for unattended home sleep studies[J]. CHEST, 2003, 123(3): 695–703. DOI:10.1378/chest.123.3.695 |
| [15] | ASHKENAZY Y, LEWKOWICZ M, LEVITAN J, et al. Discrimination of the healthy and sick cardiac autonomic nervous system by a new wavelet analysis of heartbeat intervals[J]. Fractals, 1998, 6(3): 197–203. DOI:10.1142/S0218348X98000249 |
| [16] | THURNER S, FEURSTEIN M C, TEICH M C. Multiresolution wavelet analysis of heartbeat intervals discriminates healthy patientsfrom those withcardiac pathology[J]. Physical Review Letters, 1998, 80(7): 1544. DOI:10.1103/PhysRevLett.80.1544 |
| [17] | GIL E, ORINI M, BAILON R, et al. Time-varying spectral analysiscomparison of HRV and PPG variability during tilt table test [C] // 2010 Annual International Conference of the IEEE. Buenos Aires: IEEE, 2010: 3579-3582. |
| [18] | BIANCHI A M, MENDEZ M O. Methods for heart rate variability analysis during sleep [C] // 2013 35th Annual International Conference of the IEEE. Osaka: IEEE, 2013: 6579-6582. |
| [19] | PENZEL T, KANTELHARDT J W, GROTE L, et al. Comparison of detrended fluctuation analysis and spectral analysis for heart rate variability in sleep and sleep apnea[J]. Biomedical Engineering, IEEE Transactions on bio-medical engineering, 2003, 50(10): 1143–1151. DOI:10.1109/TBME.2003.817636 |
| [20] | YU E, HE D, SU Y, et al. Feasibility analysis for pulse rate variability to replace heart rate variability of the healthy subjects [C] // 2013 IEEE International Conference on Robotics and Biomimetics. Shenzhen: IEEE, 2013: 1065-1070. |



